十脚甲殻類の環境DNAメタバーコーディングのための新たなPCRプライマーセットの開発
Development of a new set of PCR primers for eDNA metabarcoding decapod crustaceans
魚類や哺乳類を網羅的に検出するプライマーセットが開発されてきた中で、次はザリガニ・エビ・カニなどを含む十脚甲殻類を検出するためのユニバーサルプライマーの開発が課題になります。十脚甲殻類は商業的にも重要な種が多い分類群です。
十脚甲殻類に環境DNAを適用した例としては、Larson et al. 2017(ザリガニ)、Wu et al. 2018(スジエビ)などがあり、季節的なモニタリングにも利用されています。
今回開発された MiDeca-F/R(マイデカ)は、ミトコンドリアDNAの 16S rRNA 領域上に設計されています。
作成時に考慮された点として、以下が挙げられます。
- 環境DNAは断片化しやすいため、増幅産物長は200 bp未満であること。
- 増幅する領域に、種ごとに十分な塩基配列の差異があること。
- 標的種では、保存的領域(20〜30 bp)が「種を見分ける領域」の両端に存在していること。

ニホンザリガニやテナガエビ、イセエビなど、NCBIに登録されている配列
MiDeca-F/R の検証は、組織抽出DNAと実際の海水サンプルを用いて行われています。
千葉県 房総半島

採水風景
採水は、10%ブリーチでリンスした8 Lバケツを用いて10回採取し、50 mLテルモシリンジと0.45 µm孔径のステリベクスで合計1 Lをろ過しています。その後、RNAlaterを1.6 mLステリベクス内に添加し、DNAの分解を抑制しています。
RNAlaterの使用は、SIGMA-ALDRICHの製品情報を参照して適切に行いましょう。
ステリベクスからの抽出は Miya et al. 2016 を改変したプロトコルで実施しています。現在であれば、環境DNAマニュアルの手順に従えばよいと思います。概略は以下の通りです。

ステリベクスからのDNA抽出
ライブラリ調製は、以下の試薬構成と温度サイクルで行っています。
| 1st PCR(12 µL) | n = 1 | 備考 |
|---|---|---|
| KAPA HiFi Hot-Start ReadyMix | 6 µL | |
| MiDeca Primer F(5 µM) | 1.4 µL | 100 µM TE溶解液で納品→20倍希釈して使うと楽 |
| MiDeca Primer R(5 µM) | 1.4 µL | 100 µM TE溶解液で納品→20倍希釈して使うと楽 |
| 脱イオン精製水 | 1.2 µL | Milli-Q水やPCRグレード水でも可 |
| DNAサンプル | 2 µL |
1サンプルにつき8反復
| 1st PCR | 温度 : 時間 | サイクル |
|---|---|---|
| DNAの初期変性 | 95℃ : 3分 | 1 |
| DNAの変性(2本鎖→1本鎖) | 98℃ : 20秒 | 38 |
| プライマーのアニーリング | 60℃ : 15秒 | 〃 |
| 伸長反応(1本鎖→2本鎖) | 72℃ : 15秒 | 〃 |
| 最終伸長反応 | 72℃ : 5分 | 1 |
| 2nd PCR(15 µL) | n = 1 | 備考 |
|---|---|---|
| KAPA HiFi Hot-Start ReadyMix | 6 µL | |
| MiDeca Primer F(5 µM) | 1.4 µL | 100 µM TE溶解液で納品→20倍希釈して使うと楽 |
| MiDeca Primer R(5 µM) | 1.4 µL | 100 µM TE溶解液で納品→20倍希釈して使うと楽 |
| 脱イオン精製水 | 1.2 µL | Milli-Q水やPCRグレード水でも可 |
| DNAサンプル | 2 µL |
1サンプルにつき8反復
| 2nd PCR | 温度 : 時間 | サイクル |
|---|---|---|
| DNAの初期変性 | 95℃ : 3分 | 1 |
| DNAの変性(2本鎖→1本鎖) | 98℃ : 20秒 | 12 |
| プライマーのアニーリングと伸長反応(shuttle PCR) | 72℃ : 15秒 | |
| 最終伸長反応 | 72℃ : 5分 | 1 |
2nd PCR後は、電気泳動によるターゲットサイズDNAの選別と確認、濃度調整を経て、MiSeq(2×150 bp PE)で配列決定を行っています。
解析は USEARCH v10.0.240 を用いて以下のステップで行っています。USEARCHは、NGSデータの前処理から参照配列の登録までを一通り扱える解析パイプラインとして捉えると分かりやすいと思います。Web版のMiFish pipelineにも採用されています。
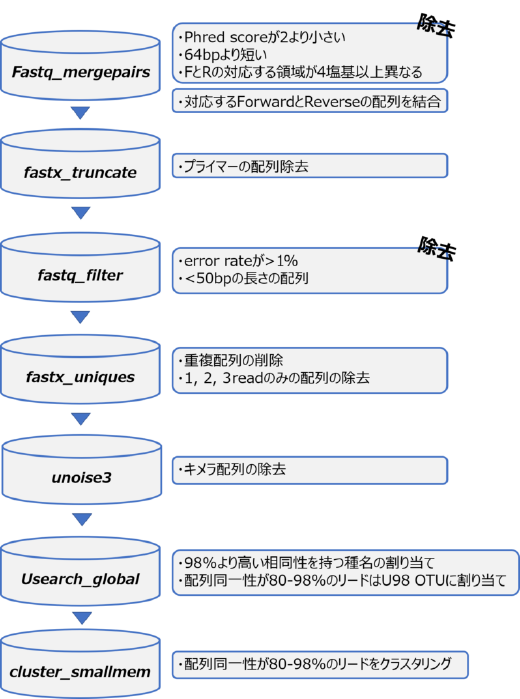
USEARCHによる解析パイプライン概要
組織DNAによる検証では、MiDeca primer で 65科・186属・250種の検出に成功しています。
野外水サンプルからは、相同性98%超で分子同定すると34種の甲殻類が検出されました。また、相同性80%以上〜98%以下では10種が同定されていました。なお、その中には生息が未確認の種も一部含まれていたとのことです。
組織片で250種の増幅が確認されていますが、日本に生息する十脚目は1054種であり、これは全体の約36%に相当します(世界では2,890種・950属・147科)。
魚類と比べると、どの分類群でも参照データベースの充実度が十分とは言い難い状況です。そのため、今まで以上に分類学的作業(同定・配列決定・登録)を進めていく必要があると言えます。
日本で作成されたユニバーサルプライマーとしては、MiFish、MiDeca、MiMammal、MiBird、Hynobius_12s などがあります。環境DNAによる生物相モニタリングを今後進めていくのであれば、これらの領域を中心にデータベース拡充を進めていくのも有効だと、個人的には考えています。
画像はジャーナルのCC BYに従い、記事で紹介したオープンアクセス論文中の画像、または自身で作成したものを使用しています。