魚類の環境DNAをメタバーコーディングするためのユニバーサルプライマー (MiFish:マイフィッシュ) の開発者本人によるレビュー論文です。長いので数パートに分けて訳します。仕事や研究の手助けになれば嬉しいです。
MiFishメタバーコーディング:環境DNAおよびその他のサンプルから複数の魚種を同時に検出するためのハイスループットなアプローチ
Introduction
生物多様性のモニタリングは、生態系の保全と生物資源の持続可能な利用に不可欠であることが、ここ30年間で広く認識されてきました。
水棲生態系から直接または間接的に大きな恩恵を受ける漁業は、利益を最大化するために単一種の漁獲量を最大化することに焦点を置き、結果として生態系構成要素の相互作用を無視することが多々ありました。
しかし現在、世界の漁業管理は、単一種に着目したアプローチから、生態系全体を視野に入れるアプローチへと転換しつつあります。
このような「生態系に基づく漁業管理 (ecosystem-based fisheries management) 」の目的は、漁業と、それを支える健全な生態系を維持することです。
生態系の健全性を評価するには、継続的なモニタリングが必須になります。というのも、環境の劣化や生物多様性の損失は、生態系に経時的な変化が生じた場合にのみ把握できるからです。
魚類モニタリングを例にすると、魚類は世界で32,000種以上が知られており、さらに2005〜2014年の間に年間平均400種の新種が記載されています。未知種を多く含むこの分類群のモニタリングは、生息環境が水中であることから調査コストが高く、分類学的専門知識も必要です。
こうした方法論的課題の解決策の一つとして、環境DNA (eDNA) 分析が挙げられます。環境DNAは、水や沈殿物などの環境試料中に存在する生物由来の遺伝物質と定義されます。
環境DNAは、一定量の水をろ過してフィルター上に捕集し、DNAを抽出します。対象分類群用に設計されたユニバーサルプライマーと、ハイスループットシーケンサー (MiSeq や iSeq など:複数サンプル・複数種由来のDNA配列を同時に解読できる機器) を組み合わせることで、複数種の同時検出が可能になります (Fig.1) 。
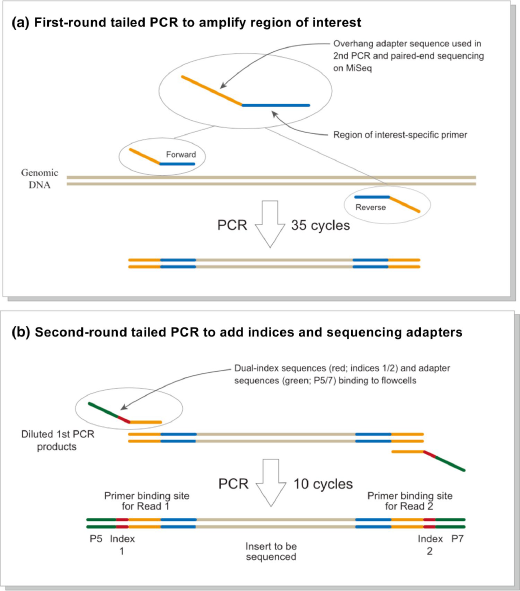
魚類を対象とするMiFishプライマーによるメタバーコーディング (ハイスループットシーケンサーによるDNA配列解読) は、世界中で実施されています。DNAqua-Net は2016年からEUの COST (CoOperation in Science and Technology) の支援を受け、次世代の水棲生物モニタリング技術の開発を目的とする、EU内外39か国からなるネットワークです。
DNAqua-Net のワーキンググループ (W3) は、DNAを用いたバイオモニタリングのフィールドおよびラボプロトコルを開発しており、2018年12月および2019年12月の DNAqua-Netニュースレター で MiFish メタバーコーディングについて議論・テストを行っています。
また、カリフォルニア海域周辺の研究では、生物多様性モニタリングの効率向上を目的に、捕獲ベースのサンプリングを補完する手法として MiFish メタバーコーディングを使用する検討が行われました。
https://edna-blog.com/paper/california-mifish/
内容の簡単な要約は、上記記事が参考になります。
日本では、「河川水辺の国勢調査」の調査法に MiFish メタバーコーディングを導入する取り組みが始まっています (Kitagawa et al. 2020) 。

環境省生物多様性センターでは、絶滅の危機に瀕している淡水魚のモニタリングに向けて MiFish メタバーコーディングの標準化を推進しています。
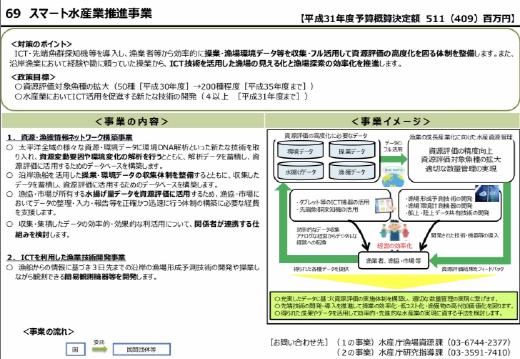
農林水産省では、今後5年間で対象となる種を50種から200種に拡大することを目標に、環境DNA分析を用いた太平洋全域の漁業資源のモニタリングを行う新たなプロジェクトを開始しました。
農林水産省では、今後5年間で対象種を50種から200種に拡大することを目標に、環境DNA分析を用いた太平洋全域の漁業資源モニタリングを行う新たなプロジェクトを開始しました。
ここからは、日本だけでなく世界中で広く使われている MiFish プライマーについてレビューしていきます。
競合するUniversal PCRプライマーとの性能比較
このセクションでは、MiFishプライマー開発の背景と、プライマー性能の in silico および in vitro 比較に関する研究について紹介します。
MiFishプライマーの開発と有用性の検証については、過去記事を参照ください。
https://edna-blog.com/paper/mifish/
MiFishプライマー配列の工夫
MiFishプライマーは、種を見分ける領域 (hypervariable region) であるミトコンドリアDNA 12S rRNA 上の 163〜185 bp (平均 172 bp) を挟むように設計されています (論文 Fig.4) 。
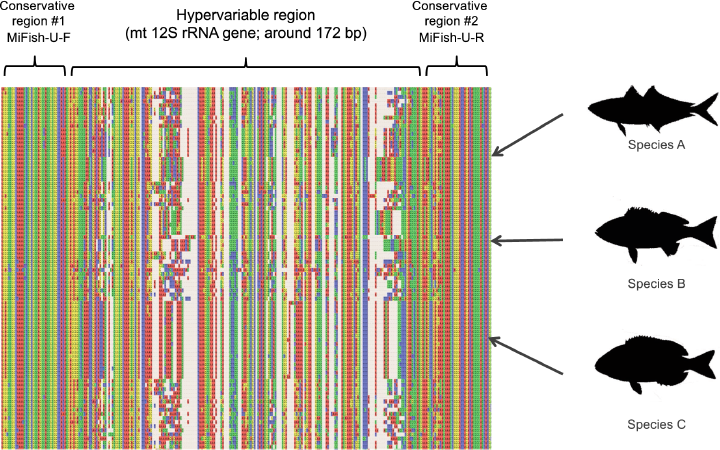
また、MiFishプライマーは縮重塩基を使わず、環境中DNAに対してアニーリングしやすくするために、以下の「技術的なコツ」を考慮して設計されています。
- プライマーの 3ʹ 末端に G/C を含める (この位置でのアニーリングが強化される)
- 3ʹ 末端の G の連続 (Gs) または C の連続 (Cs) を避ける
- T/G 結合を考慮し、プライマー領域の一塩基が C または T の場合は A ではなく G を、A または G の場合は C ではなく T を採用する
- プライマーの G/C 含有量は 40〜60%
- F/R プライマーの Tm をほぼ同一にする
これらの工夫により、プライマー領域にミスマッチがあっても、考慮しない場合より増幅しやすくなっている可能性があります。
魚類メタバーコーディングプライマーの比較 (淡水魚類群集)
MiFishプライマーに挟まれた超可変領域 (以下、種判別領域) には魚を識別するのに十分な情報がありますが、一部の種では近縁種との識別が困難な場合があります。
では、他の既存プライマーと比較した場合、分解能や検出能力はどの程度なのでしょうか。
MiFishプライマーと ecoPrimer の一つ (12SV5;Riaz et al. 2011) が増幅する種判別領域について、in silico による分類能力の比較が行われています。
種判別領域の長さは、MiFish が平均 172 bp、12SV5 が平均 106 bp です。比較の結果、MiFish の方が塩基多様性に富み、分類能力が高いことが示されました。
Bylemans et al. 2018a は、MiFish を含む既存プライマー 8 セットと新規開発プライマー 7 セットの計15セットについて、分類能力、特異性、分類範囲を評価しています。
一定の基準を満たしたミトコンドリア12S rRNA遺伝子上に設計された 3 つのプライマー (MiFish-U / Teleo / AcMDB07) を用い、人工DNAコミュニティと自然河川由来の2サンプルで 環境DNAメタバーコーディングを実施しました。
詳細は原著論文に譲りますが、結果として 3 つのプライマーペアにはそれぞれ利点と欠点があることが示されました。
その後、同流域の河川魚群集をモニタリングした実証研究 (Bylemans et al. 2018b) で MiFish が採用されていることは、MiFish の総合的パフォーマンスが優れていた可能性を示唆します。
魚類メタバーコーディングプライマーの比較 (淡水・海洋魚類群集)
英国諸島の海洋・淡水魚を対象とした研究 (Collins et al. 2019) では、4つのミトコンドリア遺伝子 (COI、cytb、12S rRNA、16S rRNA) に設計された12のプライマーセットを、参照データベースのカバレッジ、プライマーの普遍性、分類能力の観点から評価しました。
比較的高評価だった COI プライマーペア (Leray-XT、SeaDNA-mid、SeaDNA-short) と 12S プライマーペア (MiFish-U) を用いて、イギリス海峡・北海周辺の5つの河口域と沿岸サンプルで 環境DNAメタバーコーディングを行いました。
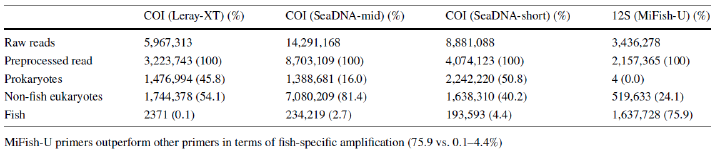
MiSeq によるシーケンスとその後の解析の結果、MiFish-U は COI プライマーと比較して、最終的に使用可能な魚リード数が最も多く、また生リードに占める魚類配列の割合も最も高い値 (COI:0.1–4.4%、MiFish:76%) を示しました (Table.1) 。
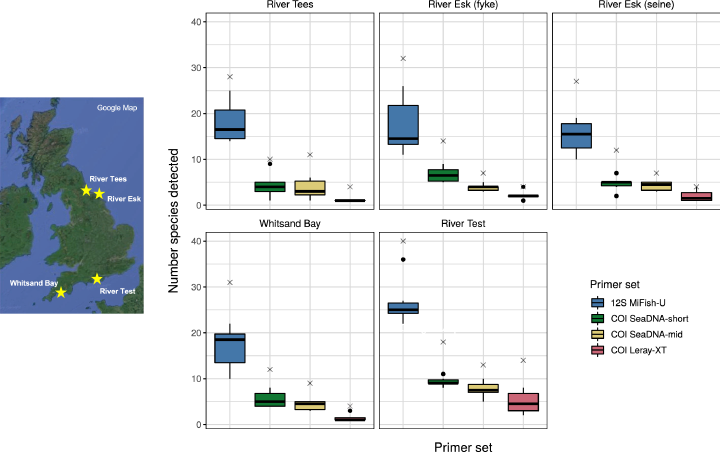
調査で生息が知られている魚種の61%しか MiFish-U の増幅領域が参照データベースに登録されていなかったにもかかわらず、種数推定はすべてのサイトで最も高い値を示しました (Fig.5) 。
また Collins et al. 2019は、縮重塩基を含む COI プライマーを用いて限定された分類群 (魚など) の 環境DNAメタバーコーディングを行うと、原核生物や非標的真核生物が増幅され、大量の「無駄な」シーケンスが生じ得ると結論づけています。
Zhang et al. 2020 は、ミトコンドリア 12S rRNA、16S rRNA、cyt b、COI を標的とした 22 のプライマーセットについて、in silico と in vitro の比較を行いました。結果として、各プライマーの増幅する分類範囲、多様性、種識別能力、群集構成は大きく異なることが示されました。
例えば北京の淡水域サンプルでは、0〜66種の魚類が検出されました。魚種を多く検出できた上位6つのプライマーセットはすべて 12S rRNA 領域に設計されたもので、そのうち MiFish を含む4セットが優れた多様性検出能力を示していました。
さらに Zhang et al. 2020 は、in silico PCRとin vitro 試験の結果が常に一致するとは限らないことも見出しています。つまり、ユニバーサルプライマーの選択は in silico 評価のみに基づくべきではないと主張しています。
その理由として、in silico 評価ではプライマー/テンプレートの不一致のみを考慮し、プライマーのアニーリングを強化する上記の技術的工夫を反映できない点が挙げられます (Ficetola et al. 2010;Elbrecht and Leese 2017;Taberlet et al. 2018) 。
以上の3つのケーススタディは、純淡水、河口域、沿岸の魚群集を対象としており、分類学的構成は地球規模で大きく異なります。それにもかかわらず、MiFish が一貫して高い性能を示したことは、このプライマーペアの汎用性を示唆します。
ただし、これらの研究は、20〜60種程度の中程度の種多様性を持つ温帯域で実施されています (Bylemans et al. 2018a;Collins et al. 2019;Zhang et al. 2020) 。
より広範な分類学的・地理的スケールで MiFish の性能を検証するには、低緯度の熱帯河川 (Cilleros et al. 2019;Sales et al. 2019) やサンゴ礁 (Oka et al. 2020) など、検出種が100種を超えることが予想される高多様性水域で評価する必要がある、と結論づけています。
和訳第一弾おわり