魚類の環境DNAをメタバーコーディングするためのユニバーサルプライマー(MiFish: マイフィッシュ)の開発者本人によるレビュー論文の和訳第二弾です。まだ数パート続きます。研究課題を考える学生さんや、新しく始める人などの仕事・研究の手助けになれば嬉しいです。
MiFishメタバーコーディング: 環境DNAおよびその他のサンプルから複数の魚種を同時に検出するためのハイスループットなアプローチ MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples
1,2章の内容はこちら
https://edna-blog.com/paper/mifish-rev1/
MiFishプライマーの分類学的最適化
MiFishプライマーが他のプライマーより優れている点が多いことは、これまでの実証研究で示されてきました。
その一方で、克服すべき課題もいくつかあります。本節では、次の2点について研究例に沿って説明していきます。
- PCRのドロップアウトによる偽陰性
- 種判別領域が複数種間で一致する問題
PCRのドロップアウトによる偽陰性について
MiFish-E / MiFish-E2
Miya et al. (2015) では、水槽内で飼育されているサメやエイなどの軟骨魚類・板鰓類(elasmobranchs)が、MiFish のオリジナルプライマーでは増幅されない事例が示されました。
著者らは、プライマーと鋳型DNAのミスマッチによるPCRバイアスが原因ではないかと考え、軟骨魚類・板鰓類(Elasmobranchs: E)でのプライミング部位の配列変異に対応するため、新しいプライマーペア MiFish-E を設計しました。
その後、このプライマーを用いることで、水槽内に飼育されている17種の硬骨魚類および板鰓類のすべてを検出することに成功しました。
しかしその後の研究により、MiFish-E ではアニーリング温度の上昇に伴い、軟骨魚類・板鰓類の検出確率が著しく低下することが明らかになりました(Fig.6;Doi et al. 2019)。
そこで、フォワード/リバースの両プライマーの 5ʹ 末端にそれぞれ1塩基を追加した MiFish-E2 を作成し、プライマーの融解温度(Tm)を上昇させることで、検出率の温度依存性を緩和しました(Miya and Sado 2019a)。
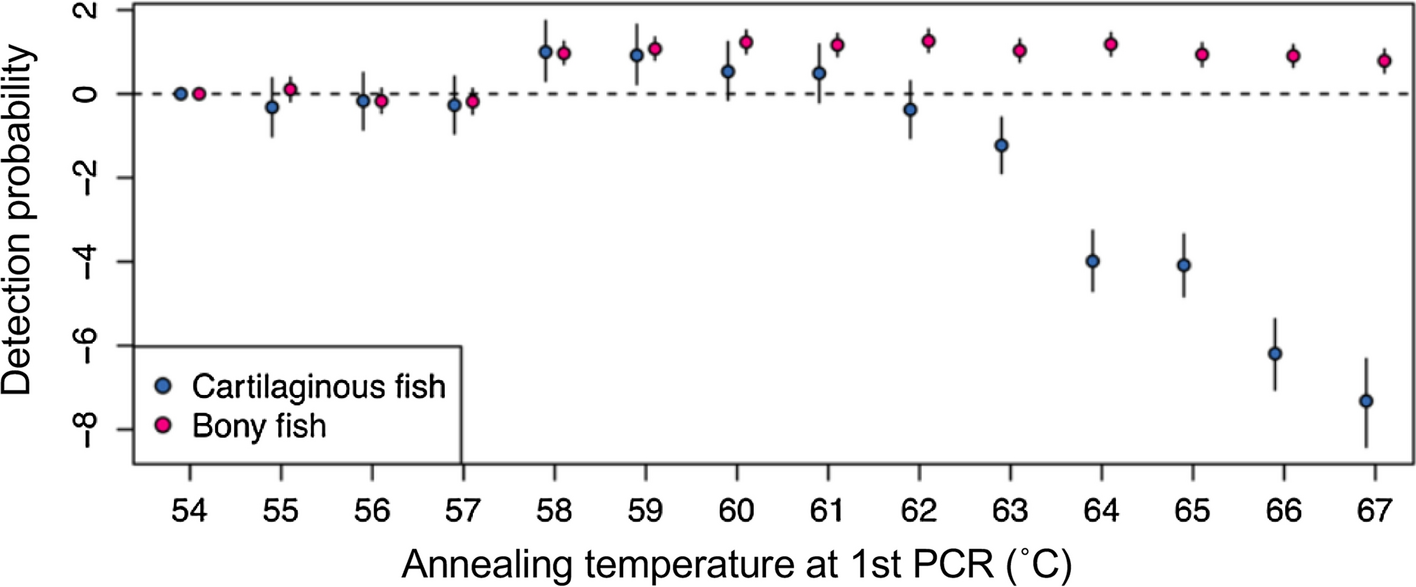
Fig.6 Doi et al. 2019 による、1st PCR のアニーリング温度に対する硬骨魚・軟骨魚の検出確率。検出確率は多種占有モデル(multispecies occupancy modeling)で推定。軟骨魚類は62℃以上で低下し、硬骨魚類は58℃以上で安定。エラーバーは95%信頼区間
MiFish-U2
日本中部の太平洋沿岸の岩礁に一般的に生息する、海のカサゴ類(4属10種: Pseudoblennius、Furcina、Ocynectes、Vellitor;Scorpaeniformes: Cottidae)は、検出されてもごくわずか、あるいは完全に検出されない場合がありました。
*
Fig.7 MiFish-U-F/R のプライミング部位の塩基配列を (a)海洋性カジカ10種 (b)キュウリウオ科4種 (c)ヤツメウナギ3種で整列。MiFish-U-R は reverse complement 表記
MiFish のプライミング部位をサンガーシークエンシングで確認したところ、MiFish-U プライマーと鋳型DNAの間に 1〜6 個のミスマッチがあることが明らかになりました(論文 Fig.7a)。
そこで、これら10種の配列変異に対応するように MiFish-U2 を開発し、その後の野外調査により、地域の魚類群集組成をより正確に反映する合理的な結果が得られました(M. Miya, unpublished data)。
著者の研究室では、海産魚類相の環境DNA増幅のために、以下の割合のプライマーミックスで 1st PCR を行ってきました(Miya and Sado 2019a)。
MiFish-U-F/R: MiFish-E-F/R: MiFish-U2-F/R = 2: 1: 1
これら2つの例は、実際の調査を開始する前に、調査海域で採取した複数の水サンプルを用いて MiFish プライマーによる予備実験を行うことが重要であることを強く示唆しています。
予備実験の結果を、捕獲による直接観察、水中での目視調査、文献による過去の生息記録などと比較することで、一般的な種でありながら結果から欠落している **明らかな偽陰性種(または種群)**を本調査前に見つけることができます。
プライマーバイアスがかかりそうなその他の種について
日本の淡水魚では、ヤツメウナギ(Lethenteron、Lampetra)や、その他いくつかの種(アユ: Plecoglossus altivelis、ワカサギ属: Hypomesus spp.)が、MiFish メタバーコーディング結果では既知の分布や個体数に比べて過小評価されている可能性が指摘されています。これはプライマーと鋳型DNAのミスマッチに起因すると考えられています(論文 Fig.7b, c)。
また、類似例として、オーストラリア・パースでのタツノオトシゴ類(Syngnathidae)に関する報告があります。Nester et al. (2020) では、新規開発プライマーではタツノオトシゴ属を検出できた一方で、MiFish を含む既存のプライマーセットでは偽陰性でした。しかしこの知見は、MiFish プライマーで2種のタツノオトシゴを検出した Tang et al. (2018) とは対照的です。
種判別領域が複数種間で一致する問題
マグロ2属について
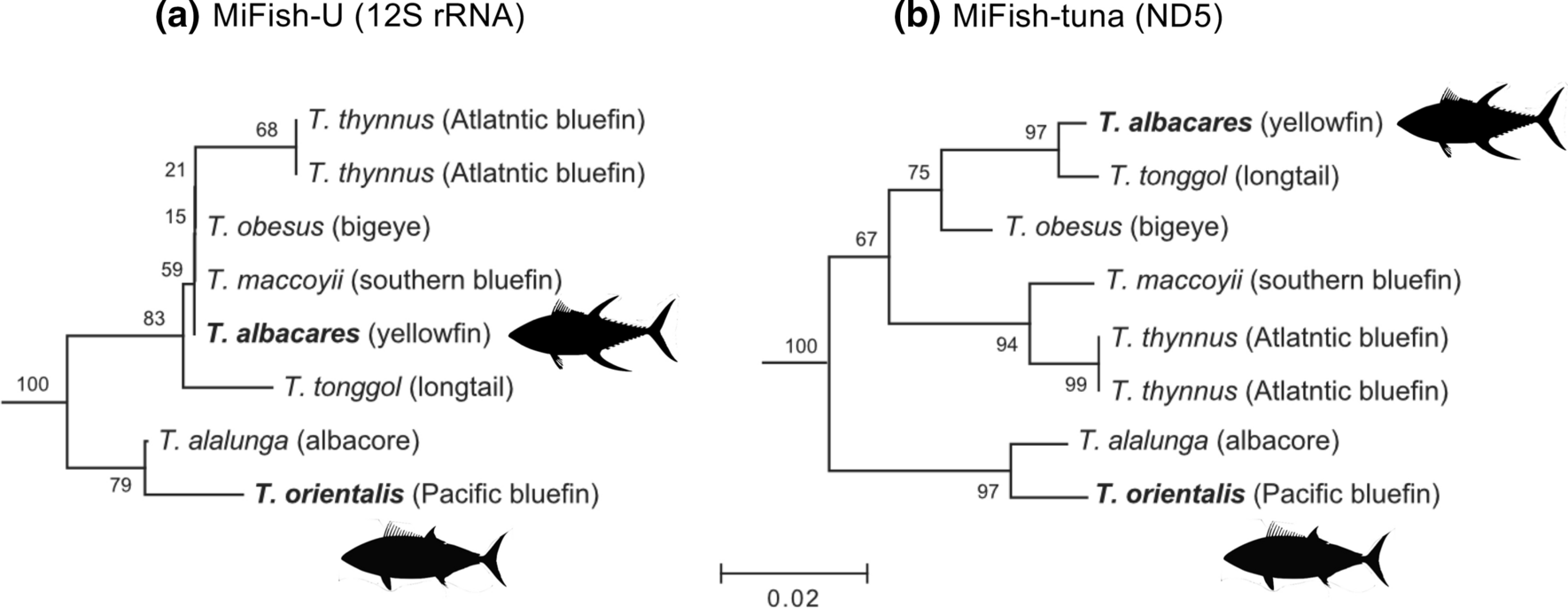
Fig.8 MiFish-U(12S rRNA領域)と MiFish-tuna(ND5領域)で増幅される領域の Neighbor-joining 法によるマグロ7種の樹形図(MEGA7)
Thunnus 属および Scombridae に属するマグロ7種では、MiFish 領域のDNA配列に、種を見分けられる十分な変異がないことが示されました(論文 Fig.8a)。
そこで著者らは、7種のミトコンドリアDNA配列を目視で比較し、構成種間で十分な種間変異を持つ ND5 遺伝子の一部(180 bp)を増幅する属特異的プライマー MiFish-tuna-F/R を設計しました。これにより、MiFish メタバーコーディングで正しく種を識別できることを確認しました。
12S と ND5 のように異なる領域に設計された MiFish プライマーを用いたマルチプレックスPCRでは、シーケンス成功のために 各プライマーの増幅領域長を揃えることが重要です。
ウナギ属について
ウナギ属(Anguilla)でも同様に、種間でDNA配列変異が乏しいことが確認されました。Takeuchi et al. (2019) は、ウナギ属16種のミトコンドリアDNA上の ATP6 遺伝子において、種間で 5〜22 塩基、3亜種間で 1〜3 塩基の差を持つ可変領域を増幅する新規プライマーセット MiEel を設計しました。
MiFish-U と MiEel を併用したメタバーコーディングでは、ニホンウナギ(Anguilla japonica)の検出に成功しました。
また Takeuchi et al. (2019) は、MiEel メタバーコーディングが、複数のウナギ属が出現すると予想される産卵場調査に有効であると主張しています。
実際、インドネシア諸島内とその周辺には8種のウナギ属が生息し、そのうち6種が南太平洋西部で産卵することが知られています。
サケ科の種
同様の例はサケ科のいくつかの種でも報告されています。Morita et al. (2019) は、ミトコンドリアDNAの ND2 領域に新規プライマーを作成しました。
シクリッド科、メバル属、トラフグ属など、ミトコンドリアのコード領域で種間差が極めて少ない種群では、環境DNAメタバーコーディングで正確に種を割り当てるため、代替的な遺伝子マーカーを見つける必要があります。
例えばタンガーニーカ湖のシクリッドでは、ミトコンドリア D-loop 領域の超可変領域を用いた新しいプライマーペアが開発されています(Doble et al. 2019)。
第二和訳おわり