魚類の環境DNAをメタバーコーディングするためのユニバーサルプライマー(MiFish : マイフィッシュ)の開発者本人によるレビュー論文の和訳第三弾です。まだ、数パート続きます。研究課題を考える学生さんや新しく始める人などの仕事や研究の手助けになれば嬉しいです。
MiFishメタバーコーディング:環境DNAおよびその他のサンプルから複数の魚種を同時に検出するためのハイスループットなアプローチ MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples
1,2章の内容はこちら
https://edna-blog.com/paper/mifish-rev1/
3章の内容はこちら
https://edna-blog.com/paper/mifish-rev2/
望ましい実験室設定と改訂された実験プロトコルについて
MiFish メタバーコーディングのための実験プロトコルは、沖縄・美ら海水族館の 4 つの水槽から採取した海水サンプルを用いて開発されました。
これらの水槽は周辺海域の魚類種多様性を再現した高密度環境であったため、サンプリングされた環境DNA濃度が高く、シーケンス用ライブラリの調整も比較的容易でした。
その後、沿岸・海洋域、深海、河川、湖沼など、緯度・標高の異なる多様な水域で実証研究が行われました。その結果、魚類の個体数密度が比較的まばらで、環境DNA濃度が不明な環境水では、実験プロトコルを最適化すべきであることが明らかになりました。
この知見を踏まえて著者らは実験プロトコルを改訂しました(例:環境DNA調査・実験マニュアル(Ver.2.2)2020年4月3日)。
また、環境DNAメタバーコーディングは外因性DNA(コンタミネーション)の影響を非常に受けやすく、DNA抽出からライブラリ作成までのあらゆる実験ステップで汚染が起こり得ます。
以下では、MiFish メタバーコーディングのための最適な実験室設定と、改訂された実験プロトコルの要点をまとめます。
実験環境について
環境DNAメタバーコーディングを成功させるための重要な課題の一つは、外因性DNAの影響を受けにくい実験環境を整えることです。
外因性DNAの供給源はさまざまに想定され、偽陽性は環境DNAメタバーコーディングに深刻な悪影響を及ぼします(Miya et al., 2015)。
外因性DNAの影響を避けるため、環境DNAメタバーコーディングを行う実験室は、作業内容に応じて以下の3室に分けておく必要があります。
- 環境DNAの抽出(Fig. 9a)
- PCRの準備(Fig. 9b)
- PCRおよびPCR後の作業(Fig. 9c)

Fig. 9 千葉県立博物館の3つの実験室を物理的に分離した例。(a)環境DNAの抽出、(b)PCRの準備、(c)PCRおよびPCR後の作業場
さらにキャリーオーバーによるコンタミネーション(汚染)を避けるため、それぞれの部屋には個別に、冷蔵庫、冷凍庫、遠心機、ラボコート、ピペット、Milli-Q ウォーターディスペンサー等が必要となります(Seffers & Schmitz, 2018)。
また、各実験ステップに専用の部屋を用意して空間的に分離することに加え、第3室(PCRおよびPCR後の作業室)から他の部屋へアンプリコン(増幅されたDNA断片)を持ち込まないよう、一方通行ルールを守る必要があります。
PCR増幅とチェックを行った後に、同日に同じ担当者が追加の環境DNA実験を行わないこと、そして使用する機器・備品は可能な限り新品(未使用)またはDNAフリーのものを使用することが推奨されます。
コンタミネーションフリーな環境DNA実験に関する注意事項は、環境DNA学会のマニュアルも参照してください。
海水サンプルのろ過法の検討
海水サンプルに対するプロトコル最適化の過程で、MiFish プライマーが時折 PCR増幅に失敗することがあります。
その最も可能性の高い理由の一つとして、試料水中に十分な量の鋳型DNAが存在しなかったことが考えられます(Miya et al., 2016)。
特定分類群の環境DNA濃度は増幅前には不明ですが、大容量の水(> 1 L)をろ過することは、外洋や深海の生態系のように魚類の豊富さやバイオマスが乏しい環境から、より多くの環境DNAを収集するための簡単で効果的な方法です。

Sterivex を用いた抽出手順例(Protocols.io)
多くの魚類環境DNA研究(Rees et al., 2014)で使用されてきたディスクフィルターに比べて、カートリッジフィルター(Sterivex©; Sigma-Aldrich, St. Louis, MO, USA)には以下の利点があります。
- 目詰まりする前に、より大きな水量に対応可能(Walsh et al., 2009)
- 個別包装で無菌
- 抽出工程など、ワークフローの一部をフィルターハウジング内で実行可能(=操作工程を閉じた系で進められる)
これらの利点により、実験室由来の汚染リスクを低減できます。
SterivexとGF/Fフィルターの比較
Miya et al. (2016) はこれらの点を踏まえ、Sterivex で水試料をろ過した後に環境DNAを抽出するプロトコルを開発し、海水水槽サンプルを用いて Sterivex と GF/F フィルターからの抽出プロトコルを比較しました。
MiFish メタバーコーディングによる解析の結果、Sterivex は GF/F フィルターよりも検出種数が有意に多いという結果が得られました(Fig. 10)。
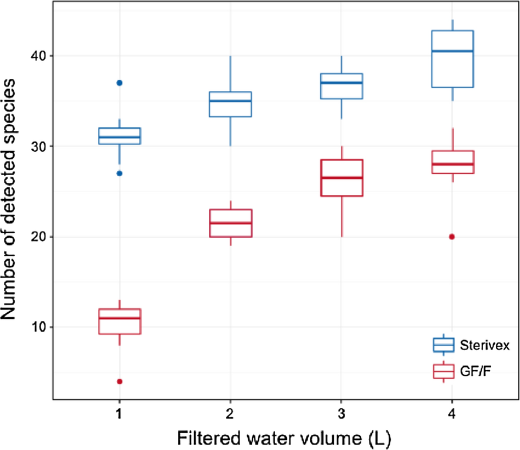
Fig. 10 沖縄美ら海水族館・黒潮水槽において、MiFish メタバーコーディングでの検出種数とろ過水量の関係を示すボックスプロット(Miya et al. (2016))。Sterivex:ステリベクス、GF/F:ガラスマイクロファイバーフィルター。
この知見は、環境DNAの捕獲効率に対するろ過法の効果を比較したその後の研究とも一致しています(Spens et al., 2016; Li et al., 2018; Capo et al., 2020)。
さらに Oka et al. (2020) はアスピレーターを用いた並列ろ過システムを開発し、14本の 1 L サンプルを同時ろ過可能で、フィルターの目詰まりが起こりにくいことを示しました(Fig. 11)。

Fig. 11 最大14本の Sterivex を同時にろ過できる手作り装置(Oka et al., 2020)。目詰まりしにくく、1 L を1時間以内にろ過可能
ライブラリ作成の失敗を避けるためのポイント(MiSeq:2段階 tailed PCR)
現在、MiSeq のペアエンドシークエンシング(増幅DNAの両端から配列解読)は、2段階の tailed PCRアプローチでライブラリを構築することが一般的です(Fig. 1)。
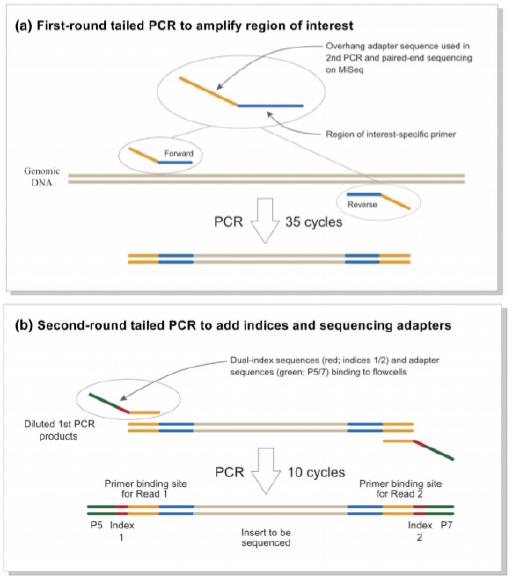
Fig. 1 MiSeq シーケンシングのための 2段階 tailed PCR によるペアエンドライブラリ調整の模式図。(a)オーバーハングアダプター配列(橙)を付加した MiFish プライマー(青)でターゲット領域を増幅する 1st PCR、(b)デュアルインデックス(赤)とシーケンシングアダプター(緑)を追加する 2nd PCR。※Miya et al. (2015) を一部改変
構築されたライブラリは、プライマーと複数のアダプター配列が両端に付加された、魚類由来の超可変領域を主成分とします(Fig. 1b)。
しかし MiSeq での配列決定に失敗した場合、プライマーとアダプターの間にあるべき MiFish 領域が欠落していたり、魚類以外の生物由来配列に置換されていたりすることがあります。
このようなライブラリ作成の失敗を避けるためには、以下が重要です。
- スピンカラムまたはビーズを用いて、1st PCR 産物を精製し、過剰なテールプライマーおよびプライマーダイマー(ハイブリダイズしたテールプライマー分子)を除去する
- BioAnalyzer / TapeStation などの自動電気泳動システムで、精製した1st PCR 産物を定量する
- 定量した 1st PCR 産物を一定濃度(例:0.1 ng/μL)に希釈し、2nd PCR のテンプレートとして使用する
- ゲル電気泳動で370 bp 付近の 2nd PCR 産物をサイズ選択し、440 bp 付近の余分なバンドを除去する
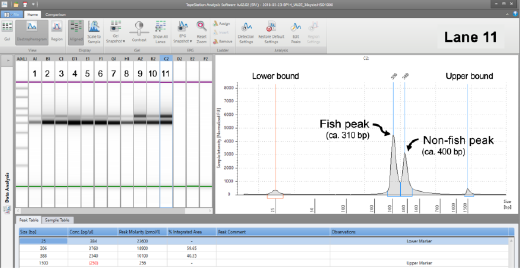
Fig. 12 Agilent 2200 TapeStation による、カラム精製後の 1st PCR 産物の電気泳動解析例。左上:11サンプルのゲル画像、右上:サンプルNo.11のピーク図、下:濃度(pg/μL)やピークモル濃度(pmol/L)などの定量結果。定量に基づき 0.1 ng/μL に希釈して 2nd PCR テンプレートとして使用する(Miya & Sado, 2019a)
Gold et al. (2020) によれば、440 bp 付近のバンドは微生物の 16S rRNA 遺伝子由来産物である可能性が示唆されています。
これらの余分なバンドは、河口や大都市近郊の湾岸部など、人為的影響を受けやすい海域でより顕著である点に注意が必要です。
上記の 4 ステップを厳密に実施することで、MiFish メタバーコーディングの高品質なデータが得られ、ほとんどの魚類について比較的一定のリード数で結果が得られるようになります。
1st PCRの繰り返し数について
環境DNAメタバーコーディングは、フィールドサンプリング、実験室実験、バイオインフォマティクスパイプラインの各段階で、種検出エラーが生じやすいことが知られています(Doi et al., 2019)。
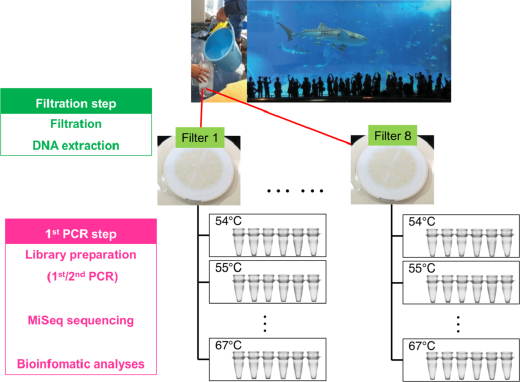
Doi et al. の研究デザイン
Doi et al. (2020)は、種組成が把握可能な水槽水を用いて、PCR アニーリング温度(プライマーがDNAに結合する温度)に伴う検出確率の変化と、ろ過および 1st PCR(同一サンプルに対する抽出・増幅の反復)における反復回数の影響を調べました。

Doi et al. より
その結果、ろ過の複製数を増やすよりも、1st PCR の複製数を増加させる方が、多種検出において有利であることが示されました。
したがって高い検出確率を維持するために、複数の 1st PCR 反復(例えば、実験操作を容易にするために 8 反復)を選択することが望ましいと考えられます(Miya & Sado, 2019a)。
この結果は、検出確率が高くない場合に少なくとも 8回反復の 1st PCR を行うことを提唱した Ficetola et al. (2015) とも一致します。
第三和訳おわり