魚類の環境DNAをメタバーコーディングするためのユニバーサルプライマー(MiFish : マイフィッシュ)の開発者本人によるレビュー論文の和訳第二弾です。まだ、数パート続きます。
研究課題を考える学生さんや新しく始める人などの仕事や研究の手助けになれば嬉しいです。
MiFishメタバーコーディング: 環境DNAおよびその他のサンプルから複数の魚種を同時に検出するためのハイスループットなアプローチ MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples
1,2章の内容はこちら
https://edna-blog.com/paper/mifish-rev1/
3章の内容はこちら
https://edna-blog.com/paper/mifish-rev2/
4章の内容はこちら
https://edna-blog.com/paper/mifish-rev3/
新しいバイオインフォマティクスパイプラインと参照データベース
MiFishメタバーコーディングでは、ハイスループットシーケンサーを用いた塩基配列決定により、一晩で数千万〜数十億のDNA配列(リード)が出力されます。
したがって、大量の生リードを前処理して解析するためには、バイオインフォマティクスパイプラインが不可欠です。
MiFishパイプラインの利点と欠点
Miya et al. 2015 は、利用可能な複数のソフトウェアを組み合わせたバイオインフォマティクスパイプラインを構築しました。
このパイプラインはその後、MitoFish サーバー上に実装され、現在はWeb上で利用できるようになっています(Sato et al. 2018)。
- MitoFish: https://mitofish.aori.u-tokyo.ac.jp/
一方で、ハイスループットシーケンサー由来のデータには、ランダムな位置で1塩基または数塩基が真の生物学的配列と異なる、多数の偽配列が含まれ得ることがよく知られています(Coissac et al. 2012、Edgar 2016)。
これらの誤りは、1st/2nd PCR、MiSeqシーケンシング時のクラスター増幅、あるいは配列解読の際に酵素が誤った塩基を取り込むことで生じる可能性があります。
さらに、MiSeqシーケンシングシステムの画像解析においても、約0.1〜1%の塩基にエラーが含まれる場合があります(Fox et al. 2014)。
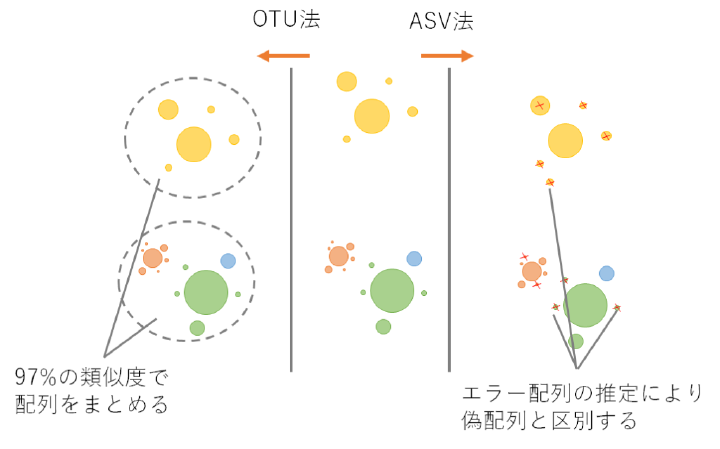
Web上で利用可能なMiFishパイプラインは、そのような誤配列を明示的に除去しない代わりに、固定の閾値(例: 97%の類似度)で類似配列をクラスタリングします。
このように、得られた配列を任意の類似度でまとめたものを OTU(Operational Taxonomic Unit)と呼び、DNA配列をOTUへクラスタリングする手法は OTU法 と呼ばれます。
しかし、OTU法の出力は再利用可能性・再現性・包括性を欠くことが指摘されています(Callahan et al. 2017)。
OTU法とは別の配列推定法(ASV法)
近年、微生物学分野では、生物学的配列をより正確に推定するために、OTU法に代わって ASV法(Amplicon Sequence Variant)が開発されました。
ASV法は、増幅や配列決定エラーが導入される前のサンプル中の生物学的配列を推定し、1塩基差の配列も区別します(Callahan et al. 2017)。
ASV法の中心となるプロセスは、真の生物学的配列は誤配列よりも繰り返し観測されやすい、という期待に基づいて両者を区別する「デノイジング」です(Tsuji et al. 2020)。
ASV法を組み込んだ新しいパイプライン(PMiFish)
我々の研究グループでは現在、このデノイジング処理をカスタムバイオインフォマティクスパイプラインに組み込んでおり、生データの前処理から、カスタムデータベースに基づく分類学的割り当てまでの一連の流れを更新したパイプラインとしてまとめています(PMiFish ver. 2.4)。
最新版は GitHub から入手できます: https://github.com/rogotoh/PMiFish
なお、PMiFishパイプラインの多くのパラメータ(例: 分類学的割り当てで用いる配列同一性の割合など)は、目的に応じてユーザーが変更できるようになっています。
MiSeqから出力されたリードの前処理と解析は、USEARCH v10.0.24(Edgar 2010)を使用し、以下の手順で実行されます。
- 品質フィルタリングしたフォワード(R1)/リバース(R2)リードのマージ(組み立て)
- プライマー配列の除去
- 品質フィルタリング
- 重複の除去
- ノイズ配列の除去(デノイジング)
- 生物種名の割り当て
ここで Edgar 2010 は、誤配列の主な原因となる シングルトン/ダブルトン/トリプレトン(全配列の中で1、2、3リードしかない配列)の除去を推奨している点に注意しましょう。
分類学的割り当ての後、PMiFish ver. 2.4 では、検出されたMiFish配列と、同一ファミリーの参照配列(カスタムデータベースに格納)を用いて、ファミリーレベルの系統樹を生成できます。
このように系統樹を視覚的に確認することで、分類学的割り当ての信憑性を検証できます。
バイオインフォマティクスパイプラインと系統に基づく確認の詳細は、Komai et al. 2019 または Oka et al. 2020 を参照してください。
参照データベースについて
Miya et al. 2015 は、環境DNAメタバーコーディングが魚類多様性調査の主流手法になる前段階として、いくつかの方法論的課題が残されていることを指摘しています。
そのうちの一つが、参照データベース(リファレンス)の完全性と正確性です。
世界中の水生環境から知られている32,000種以上の魚類を含む膨大な多様性を考えると、当時のデータベースは満足のいくものではありませんでした。
実際、カスタム参照配列データベース(“MiFish DB”)には、原著論文が発表された 2014年10月4日時点で、457科1827属に分類された約4230種の魚種をカバーする 5085本の配列が含まれていました。
その後MiFish DBは、過去5年間で着実に対象分類群を拡大し、2020年6月14日時点で登録配列数は約2倍の 9708本となりました。これは62目、479科、2766属に属する約8375種の魚類をカバーしていることになります(Fig. 13)。
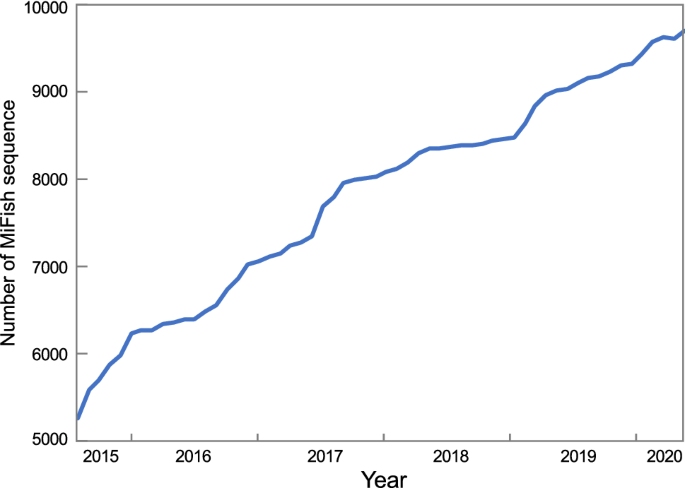
Fig. 13 Miya et al. 2015 公開後(2015〜2020年)の5年間におけるカスタムデータベース(MiFish DB ver.38)でのMiFish配列数の蓄積。
FishBase(Froese & Pauly 2019)の情報によれば、全世界の魚種数は約 34,152種で、62目、515科、5124属に分類されています。
MiFish DB ver.38 は、全世界の魚類を 目: 100% / 科: 90.3% / 属: 54.0% / 種: 24.5% 網羅しています。
日本の魚類はこれまでに 4554種が記録されており、MiFish DB ver.38 では 50目(100%)/345科(90.6%)/1302属(81.2%)/3257種(71.5%) をカバーしています。
しかし、これらのカバー率は、検出配列の正確な分類学的割り当てという観点ではまだ十分とは言えず、国際共同研究によるデータベース構築が望まれます。
この点に関連して、カリフォルニア海流域の大学・研究機関の共同イニシアティブにより、MiFishメタバーコーディングを強化する独自参照データベースが構築され、同地域の既知種864種のうち717種の参照配列が整備されていることは注目すべき点です。
参考: https://edna-blog.com/paper/california-mifish/
第4章 和訳おわり