魚類の環境DNAをメタバーコーディングするためのユニバーサルプライマー(MiFish)の開発者本人によるレビュー論文の和訳(要約)第五弾です。今回は 第6章: さまざまな水生環境での実証研究のレビューを扱います。
「実際のフィールドで MiFish がどこまで通用するのか」「環境が変わると何がボトルネックになるのか」を、海・淡水・河口(汽水)に分けて整理している章です。
MiFishメタバーコーディング: 環境DNAおよびその他のサンプルから複数の魚種を同時に検出するためのハイスループットなアプローチ MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples
- 1–2章: https://edna-blog.com/paper/mifish-rev1/
- 3章: https://edna-blog.com/paper/mifish-rev2/
- 4章: https://edna-blog.com/paper/mifish-rev3/
- 5章: https://edna-blog.com/paper/mifish-rev4/
様々な水生環境での実証研究レビュー
MiFish プライマーは水槽の 環境DNAを用いて開発された後、Miya et al. (2015) が水族館近くのサンゴ礁沿岸で自然海水を採取し、2 L x 4 サンプルから 93 種を検出しました。ここが、自然水域で MiFish eDNA メタバーコーディングの実現可能性を示した最初の例として位置づけられています。
第6章は、MiFish を実際の水域へ適用した研究を「海」「淡水」「河口(汽水)」に分けてレビューしています。実験室・水槽など高濃度で回る条件から離れるほど、(1)環境DNA濃度の不確実性、(2)混入や阻害、(3)水塊移動(流れ) といった現象が効いてきて、設計(ろ過水量、複製、陰性対照、解釈)が重要になる、という前提が敷かれています。
海の魚類群集(Marine fish community)
海域は、河川や湖沼に比べて **水塊の移動(潮汐・流れ)**が大きく、さらに塩分・水深・濁度などの環境勾配も複雑です。そのため、eDNA(環境DNA)が「その場の生物相」をどの程度反映しているか(=局所性)を読み解くには、採水設計と解釈の工夫が特に重要になります。
海洋生態系は、沿岸から外洋まで水平に広がり、地理的には極地から赤道域まで、垂直には表層から 10 km を超える深海までと、極めて多様な生息地から構成されています。本章では次の代表例が紹介されています。
Yamamoto et al., 2017は、14年に渡る長期の目視調査データがある舞鶴湾で表層・底層の計94サンプルを系統的に採水し MiFish を適用しました。結果として 128 種を検出し、目視と重なる種に加えて環境DNAによる新規記録種も得られました。採水層の選択や 1st PCR 反復数など、プロトコル最適化の重要性が示されたとまとめられています。
Andruszkiewicz et al., 2017bは、モントレー湾で空間スケールを 45 km に拡張し、深度・沿岸/深海・表層/表層下の条件を組み合わせて計 66 サンプルを解析しました。魚類 68 種を含む脊椎動物 72 種を検出し、水深帯や水塊の違いに応じた群集差を識別できる可能性が示されました。その後、カリフォルニア沖では同手法を用いた一連の研究が続きます。
Sigsgaard et al., 2019は、カタール周辺の 5 生息地タイプをカバーする 21 地点で 1 L x 3 の反復採水を行い、合計 72 サンプルを解析しました。魚類 163 種が検出され、種組成が生息地タイプを明確に反映することが示されました。種の蓄積解析からは、サンプル数の増加で多様性の大部分を捕捉できる可能性も示唆されています。
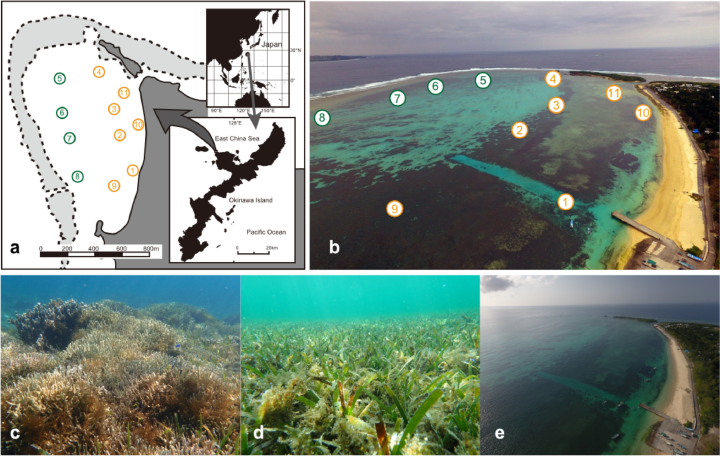
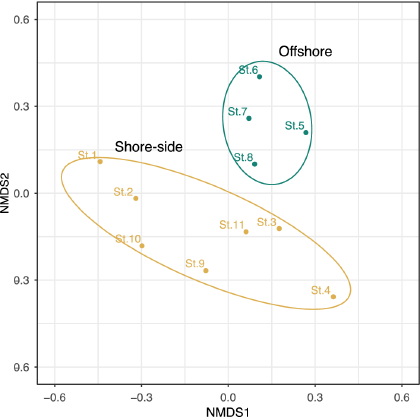
「多様性ホットスポット」での実現可能性を検証した例として、Oka et al. (2020) の沖縄島サンゴ礁ラグーンが挙げられます。捕獲調査で 217 種が確認された一方、MiFish 環境DNA メタバーコーディングでは 11 地点・1日の採水で 291 種が検出され、最終的に 410 種(119科・193属)が同定されました。両手法で共通だったのは 96 種(24%)のみで、環境DNAによる高い検出力が示されています。さらに NMDS によって、沖合のリーフ縁と沿岸の海草藻場という微小スケールの群集分化が明瞭に示されました。類似の微小スケール分化は、パルミラ環礁でも確認されています(Lafferty et al. 2020)。
Mariani et al. (2019) は、海綿動物を天然の 環境DNA サンプラーとして用い、南極・地中海の海綿試料から魚類やペンギン・アザラシ等を含む多様な分類群を検出しました。海綿自身の DNA が検出されにくい点は、プライマー/テンプレートのミスマッチの可能性として議論されています。
これら 6 つのケーススタディに加えて、インドネシア、日本、中国、欧州、北米、中南米、南アフリカなど多様な沿岸・港湾・湾・運河環境へ広く展開されており、海洋生物多様性モニタリングの適用範囲が急速に拡大していることが示されています。
淡水魚類群集(Freshwater fish community)
MiFish 環境DNA メタバーコーディングは、流水環境(河川)と止水環境(湖沼・ダム湖)の双方で実施されています。ここでは代表的な事例として、空間スケールの把握、採水戦略の最適化、**サンプル媒体(水/底質)**に関する知見が整理されています。
淡水での最初期のケースとして Nakagawa et al. (2018) は、琵琶湖周辺 51 河川・102 地点を 10 人日で調査し、既存文献の観察記録と比較しました。結果として、上流 6 km 以内の記録と 環境DNA 結果が最も整合し、上下流方向の種の入れ替わりや分布パターンの把握に 環境DNA が有効であることが示されました。
採水戦略の評価として、Bylemans et al. (2018b) はオーストラリアの河川で標高勾配に沿った 5 地点を設定し、河川横断方向に 12 本の 2 L サンプルを採取しました。その結果、河川形状・種多様性・個体数に応じて最適な採水強度が変わることが示され、今後の設計指針として位置づけられています。Sato et al. (2017) は琵琶湖の衛星湖で個別サンプルとプールサンプルを比較し、プールサンプルでは検出種数が低下したことから、種数推定には不向きだが群集の比較には有用という整理がなされています。さらに Hayami et al. (2020) はダム湖で季節変動を調べ、繁殖期に岸辺から採水することで検出効率が高まることを報告しています。
サンプル媒体の比較では、Sales et al. (2019) が南米の河川で水と底質を比較し、検出種数は概ね同等である一方、保存方法の違いで結果が変わる点を指摘しました。Sakata et al. (2020) は湖沼で水と底質を比較し、底質 環境DNA の方が分解速度が遅く濃度が高いこと、ただし種組成は大きく変わらないことを示しました。底質 環境DNA は時間スケールの異なる情報源として有望だ、という位置づけです。
捕獲法との比較では、Fujii et al. (2019)が北海道の後背湖で 7 種の捕獲調査と 環境DNA を比較し、70% 程度の種重複を報告しました。McDevitt et al. (2019) も英国の運河で電気漁法との比較を行い、環境DNA がより広い群集像を提供できると結論づけています。これらは、流水・止水の双方で 環境DNA が実用的なモニタリング手法になり得ることの実証例として紹介されています。
このほか、北米・南米・欧州・アジアの多様な河川・湖沼・運河・農業水路で MiFish メタバーコーディングが適用されており、季節成層による鉛直分布の違いなど、淡水ならではの課題にも研究が拡張されつつあります。
河口(汽水)の魚類群集(Estuarine fish community)
河口域は淡水と海水の境界で、回遊・産卵・育成など多様な魚類が集まる重要な生息地ですが、人為影響により劣化・改変されやすい環境でもあります。本章では、東アジアで近年相次いだ 3 つの研究が中心に紹介されています。
長江河口では Zhang et al. (2019) が 2018 年の 3 季節に 50 サンプルを採取し、41 種を検出しました。群集構成は季節で大きく変化し、漁業資源モニタリングへの有効性が示されたとされています。
珠江河口の沿岸湿地を対象とした Zou et al. (2020) は、環境DNAと底引き網を同時に実施し、MiFish では 57 種(24 科 47 属)、トロールでは 32 種(19 科 26 属)を検出しました。両手法を併用することで、より現実的な多様性評価が可能という結論です。
日本列島の 5 河口を広域に比較した Ahn et al. (2020) では、開発度の異なる河口で計 182 種(67 科)が検出され、河口ごとに群集構成が変わる一方、人為影響が強いほど種数が減る傾向が示されました。
補足として、英仏海峡・北海周辺の河口でのプライマー比較(Collins et al. 2019)や、河口域のエビの胃内容物を MiFish で解析して魚類多様性を推定する研究(Siegenthaler et al. 2019)など、河口域ならではの応用も紹介されています。