上記論文で使用された ASV 法を採用した、新しい MiFish パイプライン PMiFish の紹介です。
2020年12月現在の Web 版 MiFish パイプラインは OTU 法がベースですが、PMiFish はノイズ除去で利点のある UNOISE3 を用いた ASV 法と、Blast+ より高速にデータベース照合を行い合致する配列を探索できる usearch_global を採用しています。
Githubにあるチュートリアルに補足を加えながら、手順に沿って進めていきます。
はじめに
windowsを使用している場合は、WSL (Windows Subsystem for Linux) の有効化と、Microsoft StoreからUbuntuをインストールしてください。
やり方は下記に記載しています。また、仮想マシンソフトウェア VirtualBox による Linux のインストール手順も、随時追加予定です。
どこかで筆者が解析に使った覚えのあるものを引っ張り出してまとめているので、環境構築をしても無駄にはならないはず……?です。
https://edna-blog.com/analyze-env/
注意というほどではありませんが、インストールやコマンド実行の前に、全体を一度さっと確認してから進めてみてください (2021/3/20追記)。
動作環境
- Windows 10 Home
- WSL(Windows Subsystem for Linux)
- Ubuntu 20.04 LTS
- ActivePerl 5.24.3(Windows で Perl を動かすために使用)
表記ルール
コマンドラインでのコマンド表記
以下のように $ から始まる行は shell のコマンド、# はコメント行です。コマンドを Ubuntu にコピペする場合は、$ より後ろをコピーして右クリックで貼り付けます。
# ファイルやディレクトリの情報を表示するコマンド
$ ls
Ubuntu 上で、上記の $ を抜いたコマンドを実行してみてください。実行中のフォルダ(カレントディレクトリ)内にあるファイルやフォルダが一覧表示されるはずです。
出力結果
出力結果がないと、うまくいっているのか分かりにくいと思います。ここでは可能な限り、下記のような出力例も掲載します。
では始めます。
Ubuntu を起動して git clone できるようにする
いきなりチュートリアルには載っていませんが、解析のための環境を整える作業です。
解析パッケージはソースコードが GitHub にまとめられていることが多いです。ディレクトリ構成などを 1 から真似して動作環境を作成するのはミスの原因にもなり得るため、開発者が提供しているファイル構成を複製(= clone)するのが最も簡単です。
GitHub からクローンする準備をしていきます。
まず、デスクトップのアドレスバーに ubuntu.exe と入力して Enter を押すと Ubuntu が起動します(背景色は設定で変更できます)。

Gitをインストールします。
# パスワードを聞かれるので、Ubuntu をインストールした際に設定したパスワードを入力
$ sudo apt install git
Git がインストールできたら、以下のコマンドを実行して C ドライブに MiFish というフォルダを作成します。
# ディレクトリを C ドライブに移動
$ cd /mnt/c
# C ドライブに MiFish というフォルダを作る
$ mkdir MiFish
# MiFish フォルダに移動
$ cd MiFish

Git cloneする
ファイル構成を複製(= clone)します。
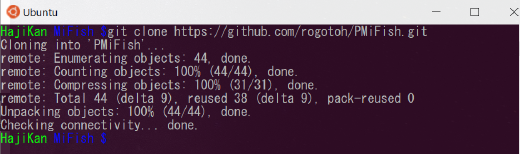
$ git clone https://github.com/rogotoh/PMiFish.git
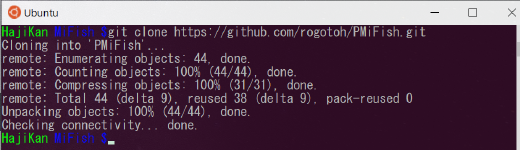

これで PMiFish を使うためのファイル一式が手に入りました。
MiFishフォルダ内に作成されたPMiFishフォルダの manual(japanese).pdf が PMiFish 2.4.1 の日本語マニュアルになります。マニュアル記載の指示に従っても設定できると思います。
使いそうな別パッケージもインストールしておく
ファイルをコマンドライン上からダウンロードするのに便利な wget や、ファイルツリーを表示できる tree などもインストールしておきます。
# パスワードを要求されるので入力する
$ sudo apt install wget tree
ActivePerl、Usearch11(, Gzip)を手に入れよう
ここからは、必要な以下のソフトウェアをダウンロードします。
- ActivePerl:Windows で Perl を動かすために必要
https://www.activestate.com/activeperl - Usearch11:解析の中核を担うパッケージ群
https://www.drive5.com/usearch/download.html - Gzip:圧縮されている Fastq ファイルの解凍・圧縮に使用
http://gnuwin32.sourceforge.net/packages/gzip.htm
ActivePerl のインストール
https://www.activestate.com/products/perl/ にアクセスすると下記画面が表示されます。

5.5 MB ほどのイメージファイルがダウンロードフォルダに保存されます。
Usearch11 の実行ファイルを入手してパスを通す
マニュアルには「Usearch11 の実行ファイルを PMiFish フォルダ内の Tools フォルダに入れ、そこにパスを通す」よう指示がありますので、その通りにします。
https://www.drive5.com/usearch/download.html にアクセスすると下記のような画面が表示されます。
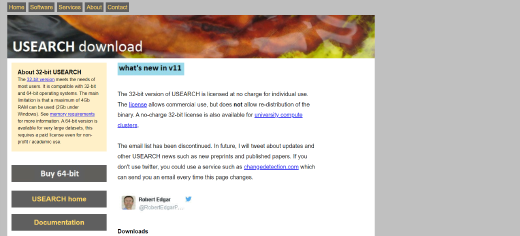
64bit 版は有償のため、ここでは 32bit 版をダウンロードします。
ダウンロードされた .gz ファイルの解凍にはフリーソフトを使用します。もしフリーソフトをダウンロードしたくない場合は、後述の「2:wget と gzip を使う」手順でダウンロードと解凍を行ってください。
wgetと(PMiFish 実行時にも使用する)gzip を使ってダウンロードと解凍を行う方法だと、4コマンド程度で完了するので簡単です。
手動でダウンロードして Lhaplus(フリーウェア)を使って解凍する
まず、Lhaplusをここ からダウンロードします。

右側の「窓の杜からダウンロード」からファイルを取得し、指示に従って Lhaplus をインストールします。
Lhaplus をインストールし終わったら Usearch のページに戻り、Windows 用のusearch11.0.667_win32.exe.gz(770 KB)をダウンロードします。
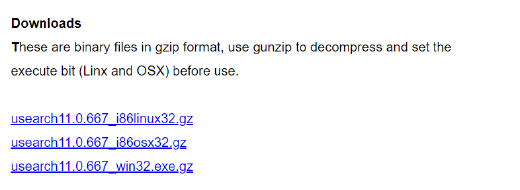
特に保存先を指定していなければ、ダウンロードフォルダに保存されます。そこで、右クリック →「プログラムから開く」→「別のプログラムを選択」→「その他のアプリ」→ Lhaplus を選択して実行します。
候補に Lhaplus がない場合は「この PC で別のアプリを探す」を押し、C:\Program Files (x86)\Lhaplus\ 内の Lhaplus.exe を指定します。
デスクトップに解凍されたフォルダが作成されるので、その中のusearch11.0.667_win32.exeをコピーし、C:\MiFish\PMiFish\Tools に貼り付けます。
以前はメールアドレス等を求められた記憶がありますが、v11 からは無いようです。
wget と gzip を使ってダウンロードと解凍をする
コマンドで Usearch の実行ファイルをダウンロードして解凍します。
現在 Ubuntu 上ではPMiFishフォルダにいる前提なので、Toolsフォルダに移動します。
$ cd Tools
wget を使ってダウンロードします。
$ wget https://www.drive5.com/downloads/usearch11.0.667_win32.exe.gz
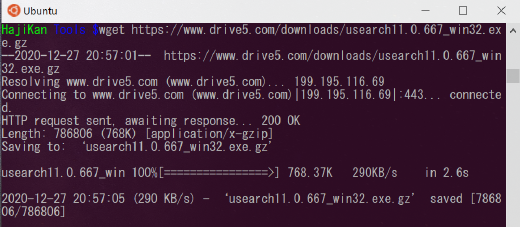
ダウンロードできているか確認します。
$ ls

gzipで解凍します。
$ gzip -d usearch11.0.667_win32.exe.gz
確認すると .gz拡張子が外れたファイルがあるはずです。これでUsearch実行ファイルのダウンロードと解凍は完了です。
# 一つ前のフォルダに戻る
$ cd ..
Tools フォルダでの作業が終わったので、1つ上の階層に戻っておきます。
Usearch11の実行ファイルのパスを通す
PMiFish は「Tools の中に Usearch11 の実行ファイルがある」前提でスクリプトが書かれているため、実行ファイルがない状態で実行すると「Tools に置いてください」というエラーが出ます。
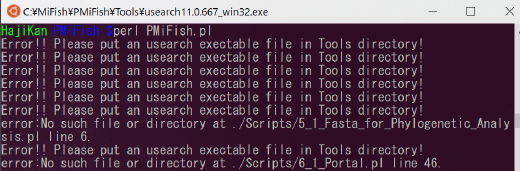
「パスを通す」とは、PC に「動かしたいソフトの場所」を教えることだと思ってください。
Windows でパスを通す簡単な方法は「ユーザー環境変数」に追記することです。ただし、ここには他のソフトの設定もあるため、必要でなければ他の部分は触らないようにしてください。
やることは、既存の書式に合わせて Usearch11 実行ファイルがあるフォルダのパスを追加するだけです。
Windows のスタートメニューから「設定」を開き、「システムの詳細設定」を開きます。
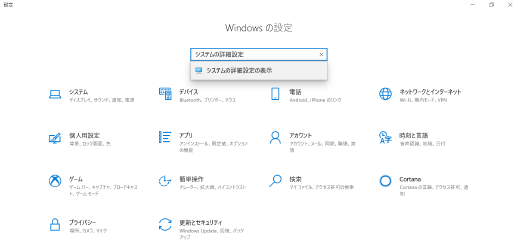
右下の「環境変数」をクリックします。
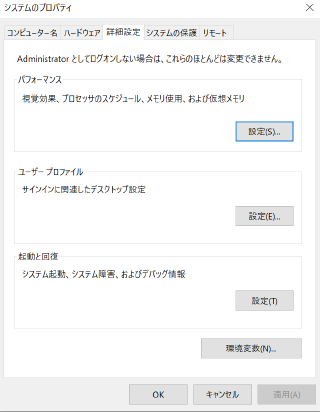
上下に同様の領域がありますが、今回は上側の「ユーザー環境変数」の中にある Path を編集します。Path をダブルクリックしてください。
「環境変数名の編集」ウィンドウが開くので、右側の「新規」をクリックし、C:\MiFish\PMiFish\Tools(バックスラッシュは環境によって “円マーク” 表示になることがあります)を追加して OK をクリックします。
これでパス設定は終了です。
参考: https://www.atmarkit.co.jp/ait/articles/1805/11/news035.html
PMiFish のフォルダ構造
上記作業が終わった PMiFish フォルダは、以下のような構成になっていれば OK です。

各フォルダの役割は以下の通りです。
- DataBase: リファレンスデータ(FASTA形式)と primer 配列を記入したファイル
- Dictionary: 日本語・英語の種名辞書データ、および科名辞書
- Results: 結果が出力されるフォルダ
- Run: 解析したい fastq ファイル/gz ファイルを入れるフォルダ
- Scripts:
- ステップごとのスクリプトが入っているフォルダ
- ステップごとに解析したい時に使用
- Tools: Usearch および MEGA の設定ファイル(.mao)を入れておくフォルダ
- PMiFish.pl: 解析を走らせるスクリプト
- PA_with_DB.pl:
- PMiFish.pl の解析後に実行することで、科レベルの系統樹を作成
- 結果は 5-2 Summary_table 内に出力される
- Setting.txt:
- 各種設定を記入するファイル
PMiFish を動かすための設定
PMiFish のパラメータ設定は Setting.txt で行います。中身は以下のようになっています。
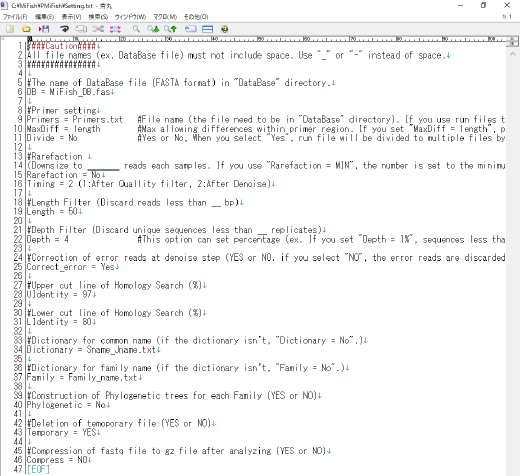
全体の編集ルールとして、以下を守ってください。
- ファイル名にスペースは使用しない(アンダーバーで代用)
#は説明文なので消さない- 半角英数のみ使用する
Setting.txt の内容は以下の通りです。基本的にはデフォルト設定でよいと思います。 オレンジで示した部分は解析に使用される部分です。
- リファレンスファイル(データベースファイルの指定)
- DataBase フォルダ内のリファレンスファイル(FASTA)を指定
DB = ファイル名- 例:DB = MiFish_DB.fas
- 使用したプライマー配列
- DataBase フォルダ内のプライマー配列ファイルを指定
- すでにプライマー配列が除去されている fastq を解析する場合は NO
Primers = ファイル名- 例:Primers = Primes.txt または Primers = NO
- プライマー領域中の最大ミスマッチ数
- 1つのプライマーペアのみ使用している場合は length 指定がおすすめ
MaxDiff = 数値またはMaxDiff = length
- 複数プライマーペアで増幅されたリードを分割するか
YESの場合、以降の解析は別々に処理Divide = YESまたはDivide = NO
- Rarefaction の設定
- リード数を希釈化する場合は数値または MIN、しない場合は NO
MINは最もリード数が少ないサンプルに合わせるRarefaction = 数値/MIN/NO
- Rarefaction 実行タイミング
- Quality filter 後:
1 - Denoise 後:
2 Timing = 1またはTiming = 2
- Quality filter 後:
- 解析に使用する配列長
Length = 数値
- 同じ配列がいくつあれば解析に使用するか
Depth = 数値またはDepth = 百分率- 例1:同じ配列が 4 以上 → Depth = 4
- 例2:各サンプルの全リード数に対する割合 → Depth = 0.01%
- Denoise でエラーとされた配列を補正して使うか
- 補正して使用:
Correct_error = YES - すべて削除:
Correct_error = NO
- 補正して使用:
- 相同性(採用する一致率)
UIdentity = 数値- 例:UIdentity = 97
- 最低相同性(指定値以下は不明とする)
LIdentity = 数値- 例:LIdentity = 80
- 標準名の辞書(Dictionary フォルダ内)
Dictionary = ファイル名またはDictionary = NO- 例:Dictionary = Sname_Jname.togodb.nonredundant.txt
- 科名辞書(Dictionary フォルダ内)
Family = ファイル名またはFamily = NO- 例:Family = Family_name_Fish.txt
- 科ごとに系統樹を作成するか(MEGAX 必要)
Phylogenetic = YESまたはPhylogenetic = NO
- 解析後に Preprocessing ファイルを削除するか
Temporary = YESまたはTemporary = NO
- fastq を圧縮するか
Compress = YESまたはCompress = NO
いくつかの変数についてのコメント
リファレンスデータベース(①)について
MiFish 領域の配列はかなりのスピードで更新されています。データベースの拡充度合いによって結果が変わるため、NCBI から独自に配列を取得する場合は、バージョン管理をしっかり行いましょう。
使用したプライマー(②)について
淡水では基本的に MiFish-U-F/R を単独で使用するか、検出しにくい種(ヤツメウナギやアユ)用にカスタムされたプライマーを併用することがあります。汽水域や海域では、アナハゼや板鰓類用の U2 や Ev2 を併用する場合もあると思います。各プライマーの長さや配列の違いによって解析結果が変わるため、使用したプライマーが正しく選択されているかを都度確認しましょう。
解析に使用する最低出現数(⑧)について
シングルトン、ダブルトン、トリプルトンは最低でも除きたいので、4 以上の数値を設定することをおすすめします。また、ハイスループットシーケンサーで得られる総リード数は都度変動するため、固定値だけではなく、全体の中での割合や近縁種とのリード関係も含めて評価するのが良いと思います。
相同性(⑩)について
MiFish の可変領域長は平均 172 bp なので、 * 1塩基差を許容: (171/172)×100 = 99.41% * 2塩基差を許容: (170/172)×100 = 98.83% のように、「1種として何塩基差を許容するか」という観点で値を決めていきます。
また、100%一致のものとそれ以外で結果を分けて出力し、後から精査を加える方法でもよいと思います。
科ごとに系統樹を作成(⑭)について
MEGAX をインストールして設定ファイルを出力した後に解析する必要がありますが、ここではその手順は掲載していません。
PMiFishを動かしてみる
ここまで来たら、あとはコマンド 1つで実行できます。まずは Setting.txt を編集せずに動かしてみます。大文字・小文字は区別されるので注意してください。
$ perl PMiFish.pl

出力された結果を見てみる
Resultsフォルダには各処理段階での出力ファイルがまとめられます。

マニュアルの 2〜3 ページにも、各出力が何を意味するかの説明があります。
ざっと結果を見るだけであれば、Portal.htmlを確認するとよいです。
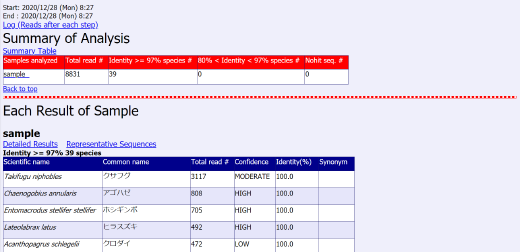
後のデータ解析や種の精査で特に必要になりやすいものは、例えば以下です(一部抜粋)。
- 2_3_Separate_chimera
- 4_1_Annotation
- 5_1_Fasta_for_Phylogenetic_Analysis
- 5_2_Summary_table
2_3_Separate_chimera
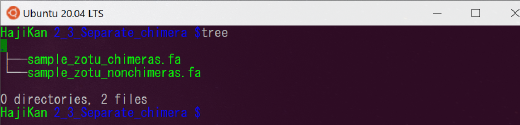
- (サンプル名)_zotu_chimeras.fa:キメラ配列が記載されたファイル
- (サンプル名)_zotu_nonchimeras.fa:キメラを除去した配列が記載されたファイル
自前のデータベース作成はできないが、最新データで相同性検索がしたい、という場合には NCBI の BLAST を使用するとよいと思います。その際は、~nonchimeras.fa を使用します。
種同定の基準は、環境省生物多様性センターの基準や精査方法に従うとよいと思います。注意点として、時折、誤同定の可能性があるデータが混じることがあるため、系統樹などを参照しながら精査を行いましょう。
BLAST 検索と結果の見方は以下の通りです。
- ここ から BLAST ページへ移動
- 上段の「ファイルを選択」から
~nonchimeras.faを選択 - 下段の「Somewhat similar sequences (blastn)」を選択
- megablast でもよいですが、データ量が極端に大きくなるわけではないので、環境DNA用途なら blastn でよいと思います
- 最下段の「Show results in a new window」にチェックを入れると、別タブで結果が表示されます
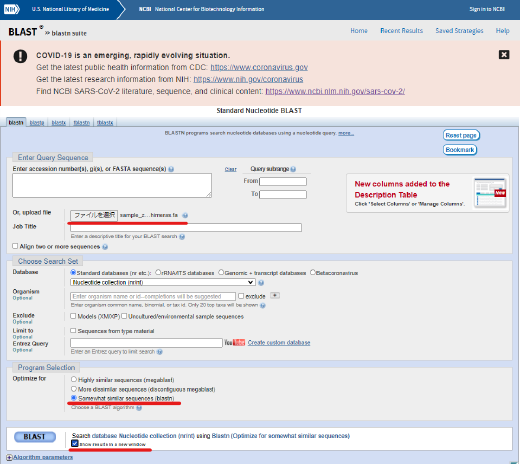
- 以下のような結果が出力されます。
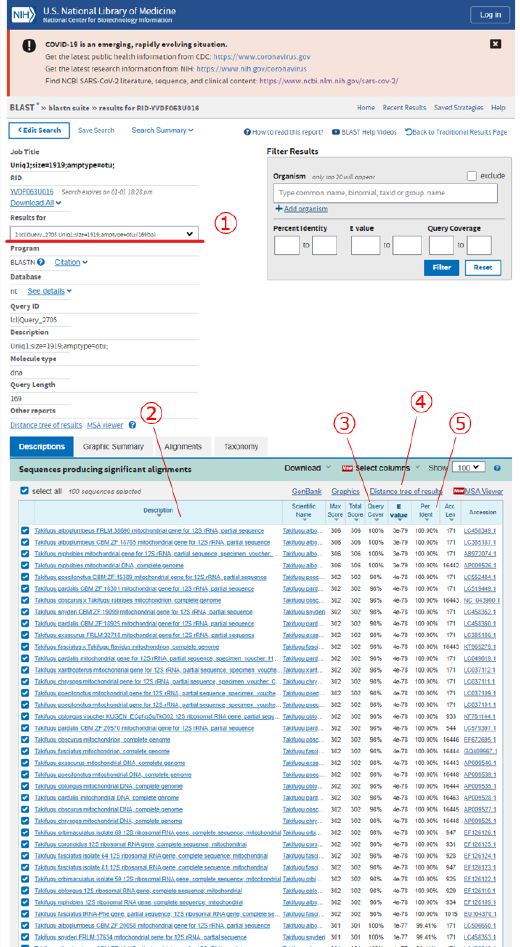
~nonchimeras.faにある配列名- ここを変更すると各配列の BLAST 結果が表示されます
- ヒットしたデータベース配列の登録種名情報
- クエリ配列の全長に対し、一致している領域の割合
- MiFish メタバーコーディングでは、ここが 100% のものを優先して選ぶべきです
- ヒット配列情報を用いて系統樹を作成
- 同じクレード内に別種が含まれないか(配列を共有していないか)などを確認します
- 相同性(どの程度一致しているか)
ただし、手作業で全サンプルを精査するのは時間・労力の面でかなり負担が大きいです。分類群の特徴が分かる人(種名を見て「怪しさ」が分かる人)が解析と精査を行うか、あるいはパイプラインのアップデートを自身で行うのがよいかもしれません。
4_1_Annotation
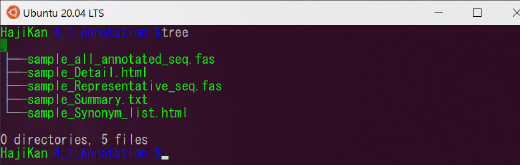
アノテーションとは、データに対して情報を付与することです。このフォルダには、パイプライン処理によって精査された配列に対して、データベースとマッチした種名やその確からしさなどの情報がまとめられています。
sample_Detail.htmlには、検出された学名、リード数、信頼度、対象配列に対して2番目に類似する種などが HTML 形式で記載されています。
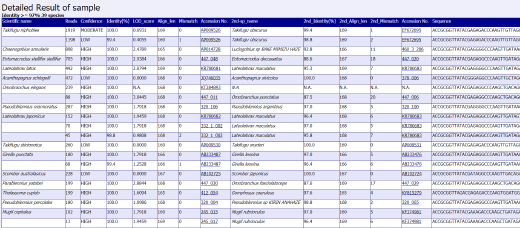
信頼度の指標として用いられているLow / Moderate / Highは Web 版 MiFish パイプラインでも表示される指標です。種精査の目安として扱います。
5_1_Fasta_for_Phylogenetic_Analysis
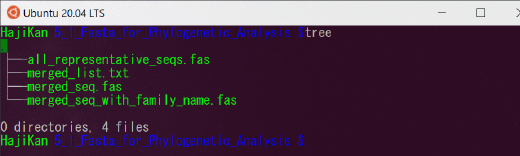
系統解析(Phylogenetic analysis)に使用する FASTA ファイルが入っています。
5_2_Summary_table
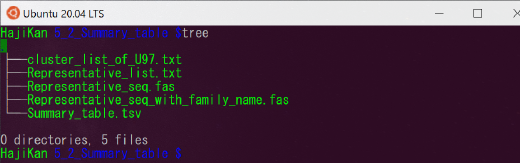
- cluster_list_of_U97.txt:Setting.txt の UIdentity を下回った配列情報(例のデータには該当がないようです)
- Representative_list.txt / Representative_seq.fas: 同定に用いた配列とその情報
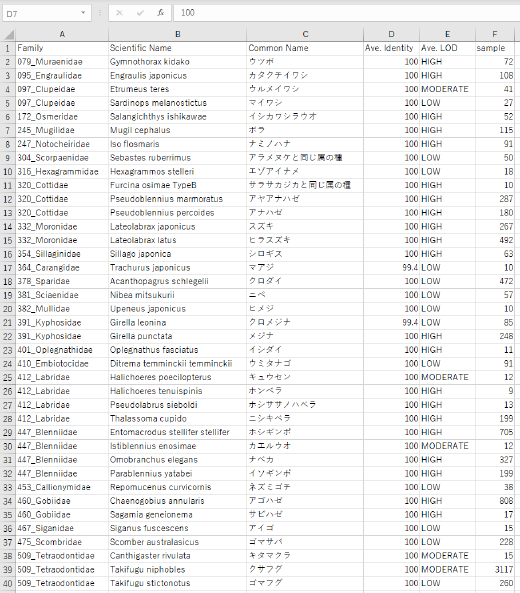
- Summary_table.tsv: 横軸にサンプル、縦軸に検出種情報を配置した表
- 4_1_Annotation の情報を、扱いやすい形式にまとめたものです
エラーについて
Q. VCOMP140.DLLが見つからない
プログラムの起動に必要なパッケージが不足している場合に発生するエラーです。
見つからないと言われたvcomp140.dll は、Visual Studio 2015 の Visual C++ 再頒布可能パッケージに含まれているとのことです。
Usearchのサイトで案内されているリンクからダウンロードします。
今回は 32bit のフリーバージョン usearch を使用しているため、32bit 版のパッケージをダウンロードします。

インストールが終わったら、エラーが出たタイミングに戻って再度実行してみてください。
Q. perl PMiFish.plと打っても実行されない
このコマンドは、「自分がいるディレクトリにある Perl スクリプトPMiFish.plを実行する」という意味です。つまり、PMiFish.plがある階層で実行しないといけません。
Ubuntu 上で現在の場所(ディレクトリ)を一度確認してみてください。
# ディレクトリ内のファイルやフォルダを確認
$ ls

# もし PMiFish.pl が無ければ cd コマンドで移動する
# MiFish フォルダにいる場合、PMiFish とだけ表示されるので、PMiFish に移動
$ cd PMiFish
# ディレクトリ内のファイルやフォルダを確認
$ ls

# 一つ前のフォルダに戻る場合の操作
$ cd ..

Q. wgetでusearchがインストールされ始めない
会社や大学環境では、プロキシサーバーによる影響が考えられます。インターネット接続自体はできている場合でも、.wgetrcにプロキシ設定(通信許可設定)を書き込む必要があることがあります。
参考 URL に従って設定するか、ネットから直接ダウンロードする方法(1)で試してみてください。
参考: https://takami-hiroki.hatenablog.com/entry/20101019/p1
Q usearchがうまく動いてなさそう
下記のようなワーニングが出る場合、Linux の言語設定(locale)が適切に設定されていない可能性があります。
perl: warning: Setting locale failed.
perl: warning: Please check that your locale settings:
LANGUAGE = (unset),
LC_ALL = (unset),
LANG = "en_US.UTF-8"
are supported and installed on your system.
perl: warning: Falling back to the standard locale ("C").
この場合、WSL の Ubuntu 環境を日本語化しておくと改善することがあります(私も同様のエラーに遭遇しました)。参考 URL の内容に従って、日本語化していきます。
# パッケージ情報の更新
$ sudo apt update
$ sudo apt upgrade
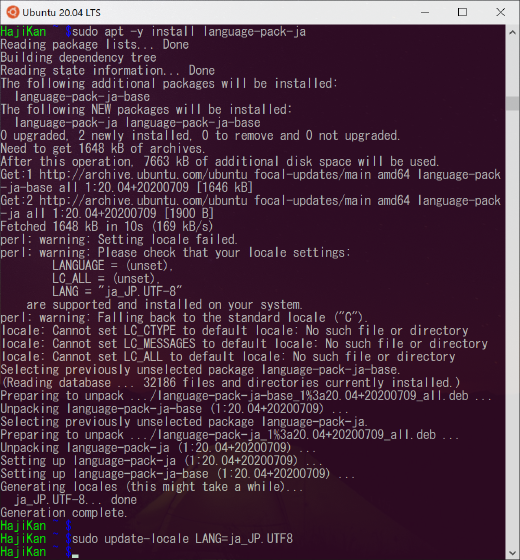
# 日本語言語パックのインストール
$ sudo apt -y install language-pack-ja
# ロケールを日本語に設定
$ sudo update-locale LANG=ja_JP.UTF8
# Ubuntu を終了して再起動
$ exit
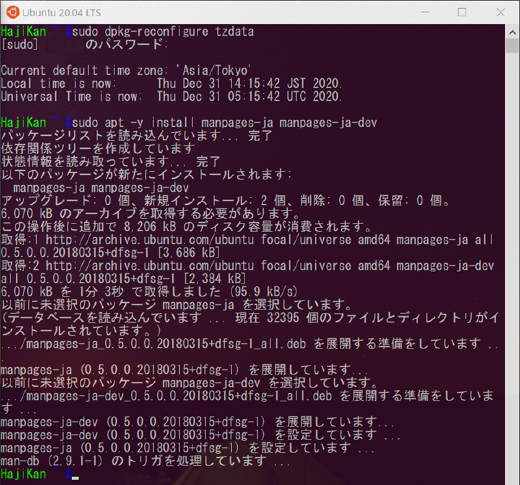
# タイムゾーンを JST に設定
$ sudo dpkg-reconfigure tzdata
# 日本語マニュアルのインストール
$ sudo apt -y install manpages-ja manpages-ja-dev
これで日本語設定になりました。
参考: https://www.atmarkit.co.jp/ait/articles/1806/28/news043.html
ちなみに、やはり英語設定の方がよい場合は、以下のように戻せます。私は日本語設定にした後、英語設定に戻しました。
# 現在の設定を確認
$ echo $LANG

# 利用可能なロケール名の一覧を取得
$ locale -a

update-localeコマンドで言語設定を変更します。
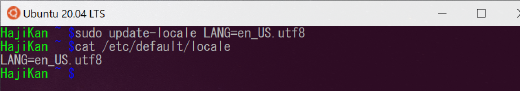
# 管理者権限で実行
$ sudo update-locale LANG=en_US.utf8
# 変更後の内容を確認
$ cat /etc/default/locale
# Ubuntu を終了して再起動
$ exit
# ubuntu を再起動後に確認
$ echo $LANG

以上、言語設定についてでした。
参考: https://www.atmarkit.co.jp/ait/articles/1610/14/news033.html
Reference
- Komai et al. 2018
Development of a new set of PCR primers for eDNA metabarcoding decapod crustaceans - Miya et al. 2020
MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples - Oka et al.2020
Environmental DNA metabarcoding for biodiversity monitoring of a highly-diverse tropical fish community in a coral-reef lagoon: Estimation of species richness and detection of habitat segregation - usearch v11
- ActivePerl
- ubuntuで(できるだけ)全部のlocaleを追加する -Qiita
- Windows10でPath環境変数を設定/編集する
免責事項
十分注意は払っていますが、本記事の情報・内容について保証するものではありません。本記事の利用や閲覧によって生じたいかなる損害についても責任を負いません。また、本記事の情報は予告なく変更される場合がありますので、あらかじめご了承ください。