淡水魚類群集の環境DNAメタバーコーディングにおけるサンプルプーリングの有用性と限界
採水の方法については、多くの方が悩むところだと思います。野外では同じ環境が再現されることはほとんどなく、採水手法を一般化することは非常に難しい問題です。
そのため、既存の論文などで検証されている方法を参考にしつつ、可能な限り妥当性の高いアプローチを選択していくことが重要だと考えられます。もし、より良いアイディアがあるのであれば、研究者と連携しながら検証を進めていくことが望ましいでしょう。
今回は、魚類を対象とした環境DNA調査において、採水方法の違いによって検出される魚類相がどのように変化するのかを検証した論文を解説します。また、自身の知識整理と考えをまとめることも目的とした書き方になっています。
淡水魚類群集の環境DNAメタバーコーディングにおけるサンプルプーリングの有用性と限界
Ficetola らが2008年に大型脊椎動物を対象とした環境DNA研究を発表してから約10年が経過した2017年の時点でも、環境DNAにおけるサンプリングデザインの最適解はまだ確立されていない状況でした。
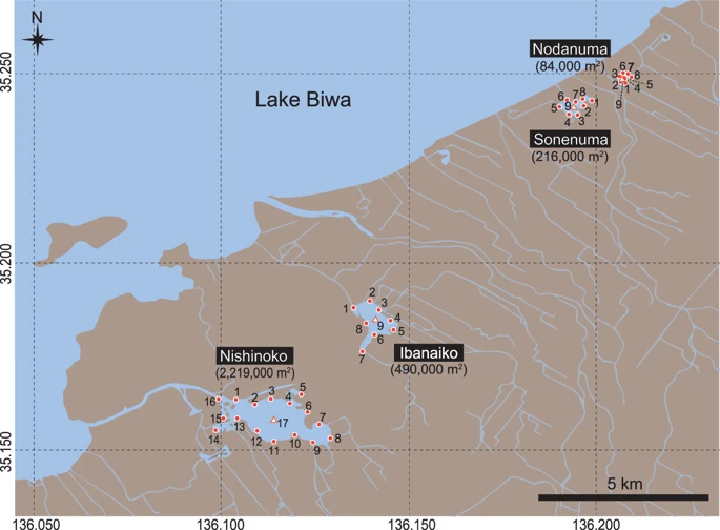
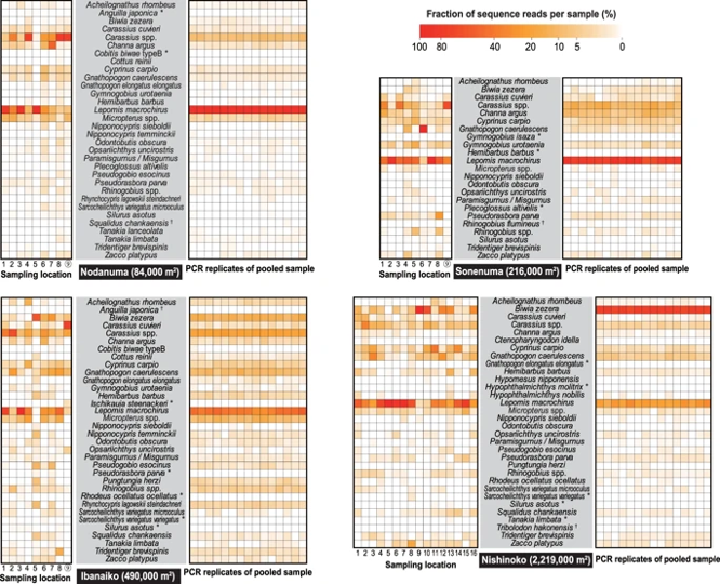
本研究では、MiFish プライマーを用いた環境DNAメタバーコーディングにおいて、琵琶湖周辺に点在する4つの内湖(野田沼、曽根沼、伊庭内湖、西の湖)の沿岸部複数地点で採水を行い、
- 各地点のサンプルを個別に分析する方法
- 同一湖内のサンプルを1つにプールして分析する方法
の2通りを比較し、検出される魚類の種組成の違いを評価しています。
メソッド
- 採水
- 採水間隔
- 野田沼:65 m
- 曽根沼:130 m
- 伊庭内湖:230 m
- 西の湖:580 m
- 個別サンプル:各地点 500 mL
- プールサンプル:同一地点で 250 mL ずつ採水し、まとめてプール
- ろ過:GF/F フィルター(0.7 μm)を用いた現地ろ過
- 採水間隔
- DNA抽出
- Yamanaka et al. (2016) に準拠したスピンカラム法
- 精製:DNeasy Blood and Tissue Kit
- 溶出量:100 μL
- 解析
- 現行の web MiFish pipeline と同様の解析フロー
- 群集解析:R の vegan、pheatmap パッケージを使用
結果と考察
個別に分析した場合、検出された魚類の種数は以下の通りでした。
- 野田沼:31 種
- 曽根沼:22 種
- 伊庭内湖:33 種
- 西の湖:31 種
一方で、サンプル間には中程度の空間自己相関が検出されました。
それに対し、湖ごとにサンプルをプールして分析した場合、1st PCR の繰り返し数を15回に設定すると、
- 野田沼:30 種
- 曽根沼:20 種
- 伊庭内湖:29 種
- 西の湖:27 種
の魚類が検出されました。
Note
※空間自己相関とは、一言でいうと、近しいものは似ていて遠いものは異なる性質のことです。 ある地点で魚採りをした場合に50cm離れた所と10m離れた所では、採れた魚の種構成は50cm横より10m横の方が違ってくるはずです。
環境と生物の種組成の違いなど、比較研究における調査設計において、空間自己相関は常に気を付けておく必要があると言えます。
下図は種数累積曲線です。サンプリング順序をランダム化して種数を累積することで、調査地点に生息する推定種数を評価します。
- 縦軸:検出種数
- 横軸:
- 個別サンプル(Individual):サンプル数
- プールサンプル(Pooled):1st PCR の繰り返し数
- 青・赤の影:95% 信頼区間
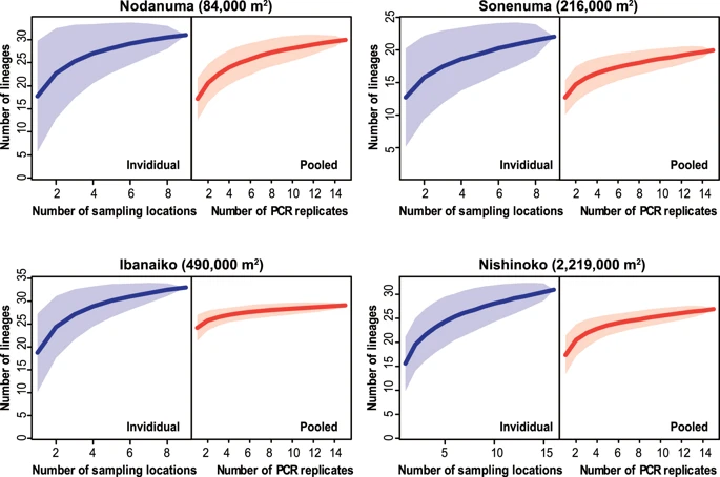
累積曲線が定常状態に近づいていれば、今回のサンプリングで検出可能な種数をおおよそ網羅できていると解釈できます。
野田沼(左上)では、両サンプリング法とも定常状態に近い形状を示しており、さらなるサンプル数の増加によっても検出種数の増加はあまり期待できないと考えられます。
一方、伊庭内湖(左下)では、プールサンプルの累積曲線は定常状態に近いものの、個別サンプルではまだ増加傾向が見られます。この差は、両手法の検出能力の違いを反映している可能性があります。
また、信頼区間(影の部分)の幅は、どの湖においても個別サンプルの方が大きいことから、個別サンプルでは1サンプルあたりの検出種数にばらつきが大きいことが分かります。複数地点のサンプルをプールし、1st PCR の繰り返しを設ける方法の方が、検出される種数と種組成がより安定していると言えるでしょう。
一般的に 1st PCR は 4 回程度の繰り返しで行われることが多いため、プールサンプルの 4 繰り返しが、個別サンプル何地点分に相当するのかを示した図を作成しました。
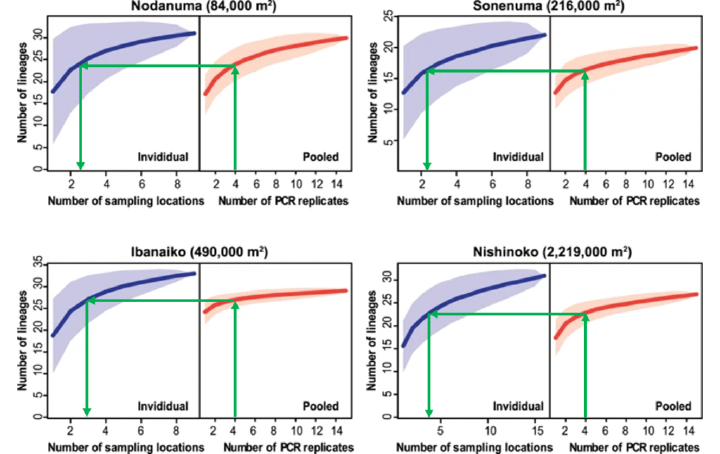
今回の結果からは、プールサンプルの 1st PCR 4 繰り返しは、個別サンプルを少なくとも 2 地点以上分析するのと同程度と解釈できます。特に西の湖のような面積の大きな湖では、複数地点をまとめて分析することで、コストを抑えつつ一定数の種を検出できる可能性があります。
ただし、それでも全個別サンプルで検出された種数の約 4/5 程度にとどまっており、プールサンプルのみで調査地の魚類相を十分に反映できるとは言い難い結果です。検出可能な限り多くの種を把握し、捕獲調査では得られない種の検出を目指すのであれば、複数地点を個別に分析する手法が依然として有効だと考えられます。
サンプリング手法間の特徴として、個別サンプルで総read数が0.05%未満の稀な種は、プールサンプルからほとんど検出されませんでした。これは、DNA 量の少ない希少種が、プールによって検出されにくくなる可能性を示しています。
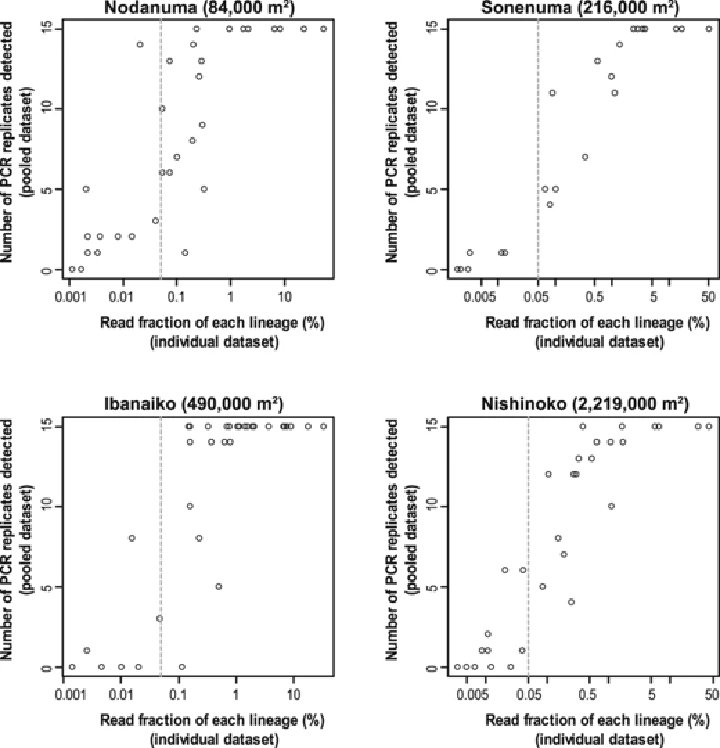
さらに、プールサンプルにおける PCR 繰り返し間では、検出される魚類の種組成が非常に類似していました。
下図は NMDS(non-metric multidimensional scaling)による解析結果です。これは、地点間の群集類似性(β多様性)を可視化する手法です。(Rを使った動かし方はこちらのブログが参考になります)
上段は個別サンプル、下段はプールサンプルの結果を示しています。
近ければ似ていて、離れていれば似ていないといった見方をします。下図上段は個別分析の魚類相のデータに対して、下段はプールサンプルについて解析を行っています。
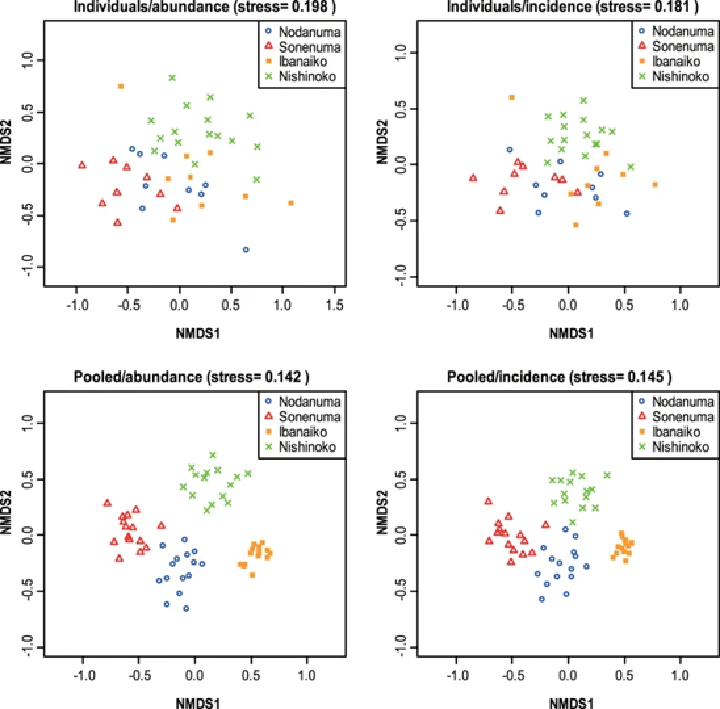
個別サンプルに比べ、プールサンプルでは各内湖ごとに点が集まっており、それぞれの内湖の魚類相の特徴がより明瞭に表れていることが分かります。
このことから、サンプルをプールして分析する方法は、多様性の最大検出には不向きである一方、調査地点間の種組成の特徴を比較する目的には有用である可能性が示唆されます。
まとめ
- 同一地点のサンプルをプールして PCR の繰り返し数を増やすよりも、個別サンプル数を増やす方が多くの種を検出できる
- 調査地点間の特徴比較には、複数地点のサンプルをプールする方法が有効である可能性がある
- Read 数の少ない(DNA 量の少ない)稀な種は、プールサンプルでは検出されにくい
- 採水地点が近すぎると、空間自己相関が発生する
(…あくまで「可能性がある」として解釈する必要があります)
※画像は、記事中で紹介したオープンアクセス論文の CC BY ライセンスに基づくもの、または自身で作成したものを使用しています。