Sterivexフィルターを使用した環境DNAの抽出・精製・測定のための改良されたプロトコルの野外適用 Field application of an improved protocol for environmental DNA extraction, purification, and measurement using Sterivex filter
先日開催された土木研究所主催の共同研究報告会にて、本論文を初めて知りました。 技術的に非常に興味深い内容であったため、ここで解説したいと思います。
本論文では、 Sterivexフィルターからの環境DNA抽出・精製プロトコルの再検討(Part1) と、 PCR阻害への対処(Part2) について検討されています。本記事(Part1)が、環境DNAの検出感度や検出種数に疑問を持っている方々の一助となれば幸いです。
(内容が長くなったため、2本立ての記事とします)
論文の要約
- ステリべクスを使った抽出法で既存の確立された方法と比較して、環境DNAの収量を12-16倍に改善
- 抽出と精製効率を回収率として評価するためにサンプルに内部標準DNAを添加
- タンパク質ベースの添加剤を使用して、試薬にProbe qPCR Mix(Takara)を使用すると阻害がかかるようなサンプルでも分析の堅牢性が向上(TaqMan Environmental MasterMix 2.0とも比較)
- さらに阻害がかかるようなサンプルにはDNeasy PowerClean Pro Cleanup Kitによる精製が必要な場合もあったが思ったより回収率は低かった(平均 33%)
抽出およびPCR阻害についての基礎的な説明
一般的な環境DNAの抽出方法
環境DNAマニュアル v2.2では、以下の2種類のろ過法が紹介されています。
これに伴い、抽出法も以下のように複数存在します。
- サリベットやスピンカラムを用いたGF/Fフィルターからの抽出
- Sterivexから直接抽出する方法
いずれの方法にも長所・短所があるため、マニュアルには複数の手法が併記されています。

サリベットを使ったGF/Fフィルターからの抽出イメージ
サリベットは15mLチューブサイズであるため、1サンプルを複数枚のフィルターでろ過した場合でも、1本のサリベットで処理できる利点があります。一方で、マイクロチューブ用ローターでは遠心できないため、大型ローターが必要となり、1回あたりの処理サンプル数はスピンカラム法より少なくなります。
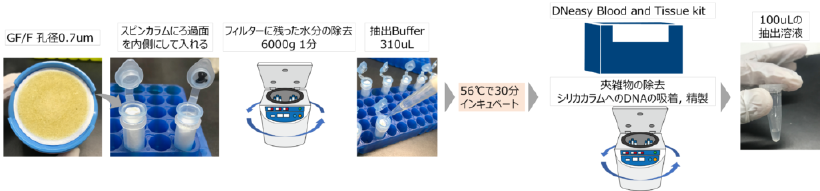
スピンカラムを使ったGF/Fフィルターからの抽出イメージ
スピンカラムは2mLチューブサイズであるため、一般的なマイクロチューブローターで処理可能です。そのため、1回あたりに処理できるサンプル数は比較的多くなります。ただし、1sンプルが複数枚のフィルターに分かれる場合、複数チューブを使用する必要があります。
ちなみに私は普段この方法を使っています。
SterivexフィルターはGF/Fとは素材や孔径が異なります。
孔径は主に以下の2種類です。
- 0.45 µm:PVDF(ポリフッ化ビニリデン)
- 0.22 µm:PVDF + PES(ポリエーテルスルホン)
フィルター素材の特性については、Cytivaのページが参考になります。

ステリべクスからのDNA抽出イメージ
マニュアルに記載されていない抽出法
千葉県立博物館の宮先生らのグループによる論文(Kawato et al.2021) では、マニュアルとは異なる方法で Sterivex からDNAを抽出しています。

Kawato et al.2021 Graphical abstractより
Sterivex カートリッジを分解し、フィルターを細断して処理する方法は、海外の研究では比較的よく見られます。
この方法では、
- フィルターを分割して処理
- 通常 440 µL の溶出量を 倍量以上 に増加
- 最終溶出量を 200 µL
といった点が、マニュアル記載法と異なります。
抽出効率は向上するものの、Sterivex の分解はコンタミネーションリスクが高くなるため、筆者自身は慎重な姿勢を取っていました。
環境DNA抽出は、PCRと異なり 同一サンプルでのやり直しができない工程であるため、最も注意が必要なステップです。
筆者が抽出工程で重要だと考えている点は以下です。
- 操作が煩雑でなく、コンタミネーションリスクが低い
- 一般的な分子生物学実験機器で作業可能
- 多数サンプル(12サンプル以上)を一度に処理可能
- 汎用的な試薬を使用
- 一部工程の自動化が可能
これらが満たされない場合、環境DNA解析の利点である「大量サンプル処理」が活かせなくなる可能性があります。
PCR阻害とは?
PCR(ポリメラーゼ連鎖反応)は、DNAを指数関数的に増幅する反応です。基本的な仕組みについては M-hub の解説が分かりやすいです。
PCRではポリメラーゼ(DNA合成酵素)を使用しますが、この活性が直接的または間接的に阻害される現象を PCR阻害 と呼びます。
環境DNA解析で問題となる阻害物質としては、
- フミン酸・フルボ酸
- タンニン酸
- ヘマチン(血液由来)
などが知られています。これらは自然環境中にも普遍的に存在するため、阻害体制の高いPCRプロトコルが不可欠です。
Uchii et al.2019では環境DNA分析の検出と定量化のためのPCR試薬の阻害体制の評価をしています。フミン酸、フルボ酸、タンニン酸に対して、以下のPCR試薬の阻害耐性評価をしています(選択された理由も記載)。
- TaqMan Gene Expression Master Mix(GMM)
- 一般的な環境DNAの研究で使用されているため
- TaqMan Environmental Master Mix 2.0(EMM)
- 一般的な環境DNA研究で使用されており、PCR阻害に対して耐性が高い(Jane et al.2015)
- TaqMan Fast Advanced Master Mix(FMM)
- いくつかの環境DNA研究で使用されているため(Uchii et al.2017, etc…)
- TaqPath qPCR Master Mix, CG (TMM)
- PCR阻害剤耐性が高く、キャリーオーバーコンタミネーションを防止するための(UNG)が含まれているため(UNGを含まないEMMより用途としてはいい?)
- KAPA3G Plant PCR Kit (K3G)
- リアルタイムPCR用には設計されていないが、植物由来の阻害に高い耐性を持つポリメラーゼが含まれているため
- Probe qPCR Mix (PQM)
- PCR阻害耐性が向上し、上記5つとは異なるポリメラーゼを使用しているため
評価の結果、
- 阻害耐性試薬でないGMM, FMMは阻害耐性試薬と比較して、フミン酸とタンニン酸に対する耐性が低かった
- 過去の研究で阻害が確認されているフィールドサンプルに対して阻害耐性のあるいくつかの酵素を使って検出を行ったところ結果が改善した。
- 阻害耐性試薬もそれぞれ得意不得意がある といった感じです。
Minegishi et al.2019は本記事の論文と同チームの人が研究に取り組んでおり、やはりこちらでもPCR阻害に関する対処が必要だったみたいです。
DNeasy Blood and Tissue kitで抽出しただけでは阻害が強いため、Zymo Research OneStep PCR Inhibitor Removal kitとDNeasy PowerClean Cleanup Kitのそれぞれで精製し、希釈とポリメラーゼを工夫したり、BSAを添加するなどして、阻害のかかるサンプルに対して最適なプロトコルを見つけています。

Minegishi et al.2019 Table1より
上記2本の論文でもわかる通り、環境DNA分析で問題となるPCR阻害はかなり厄介でかつ、統一した対処法がまだ確立されていません。
私が普段行っているのはDNeasy Blood and Tissue kitで抽出したDNA溶液をAmpureで精製する方法です。多数のサンプルを一気に処理できる点や、NGSの処理工程で使うので精製キットを追加で購入する必要もなく濃縮もかけれたりするので多用しています。
では、論文の本筋に移りたいと思います。
やったこと
本論文では、以下の2点を検証しています。
- 水槽水を用いた環境DNAの抽出・精製効率の評価
- 改良したプロトコルのフィールドサンプルへの適用
水槽実験による抽出と精製効率の検証
水サンプルのろ過
実験系はFig.1を参考にすると分かりやすいです。ステリべクス(孔径0.45μm : Millipore SVHV010RS)で水槽水をそのままと10倍に希釈したものをれぞれ1L吸引ろ過しています。ろ過後はマニュアルに記載通り1.6mLのRNAlaterをインレットから注入して-20℃で保管しました。
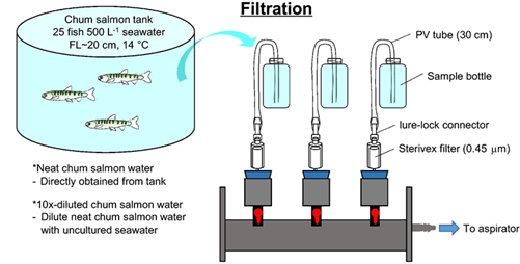
Fig.7を改変
環境DNAの抽出と精製
ステリべクスに添加したRNAlaterを解凍したのち、Outletから吸引して取り出しています。
抽出は以下の3通りの方法を試しています。
- 環境DNAマニュアル記載法
- 環境DNAマニュアルの約4倍の溶出バッファーをインレット側から添加
- 環境DNAマニュアルの約4倍の溶出バッファーをアウトレット側から添加 インキュベートはどのプロトコルも56℃で30分です。
インキュベート後はステリベクスのOutlet側を下に向けて、50mLチューブに取り付けられたスピンカラム(maxi spin)の上?にステリベクスをセットして、2500gで10分間遠心して抽出液を取り出しています。
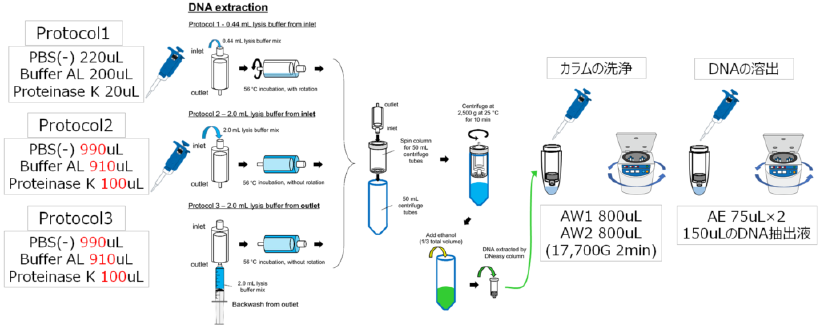
Fig.7を改変
遠心で取り出した抽出液にProtocol1は環境DNAマニュアル通りの200uL, Protocol2,3は最終容量の1/3である1mLの99.5%エタノールを添加して、DNeasy Blood and Tissue kitに付属のカラムに吸引ろ過しています。
そのあとAW1とAW2でカラムに残る夾雑物を遠心ろ過洗浄し、最後に75uLのAE Bufferを2回カラムに通して計150uLのDNA抽出液を作成しました。
抽出液はQiagen DNeasy PowerClean Pro Cleanup Kitで精製しています。
また、抽出効率のテストのために同じステリべクスカートリッジを3回抽出しています。
各プロトコル比較の結果
下記図はそれぞれのプロトコルで3回連続で抽出した抽出液をシロサケの検出系で定量PCRした時の定量値です。左からProtocl1, 2, 3となってます。

Fig.1 を改変
Protocol1(環境DNAに記載のプロトコル)は1,2回目ともほぼ同様の収量で、3回目は1回目の約1/3の収量でした(上記図A)。
Protocol2(インレットから抽出液を2mL注入)では、1回目がProtocol1と比較して、27倍の収量で、2,3回目はProtocol1と同様の収量でした。
Protocol3(アウトレットから抽出液を逆噴射)では、1回目がProtocol1と比較して、16倍の収量でしたがProtoclo2と比較すると43%少なかったです。2回目はProtocol2の2回目と比較して2倍の収量が得られました。
各プロトコルの抽出液の総DNA量を定量すると、
- Protocol1 : 189.3 ± 112.0 ng
- Protocol2 : 1183.3 ± 112.0 ng
- Protocol3 : 1183.3 ± 86.6 ng となっており、明らかにマニュアル記載のプロトコルよりDNAの収量が多いようです。
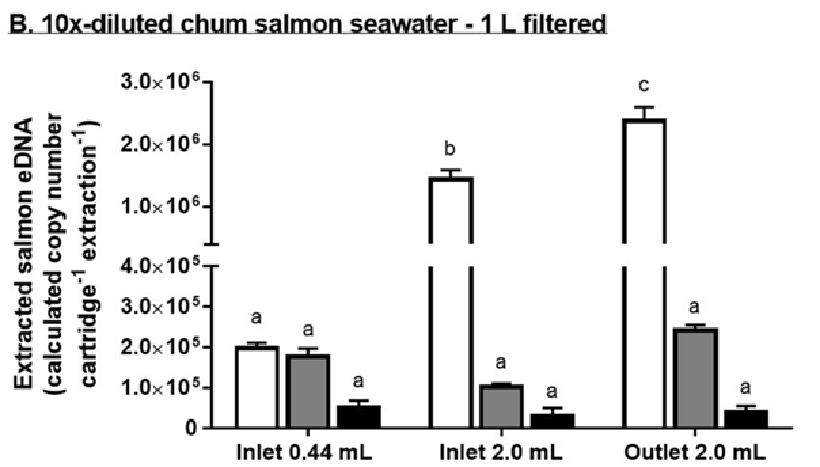
Fig.1を改変
水槽水を10倍に希釈して濾過した処理系では、そのままろ過した実験系で得られた環境DNAの収量と比較してProtocol1, 2, 3はそれぞれ、4.8%、1.3%、および3.8%でした。
Protocol1の1,2回目の抽出ではほぼ同様の収量で3回目は1/4の量まで減りました。
Protocol2では1回目の環境DNAの収量がProtocol1の7倍の収量でしたが、2,3回目はProtocol1の半分程度となりました。
Protocol3では1回目の環境DNAの収量が有意に高く、2,3回目はProtocol1と同様でした。
各プロトコルの抽出液の総DNA量を定量すると、
- 392.0±33.2 ng
- 401.3±37.8 ng
- 682.0±35.5 ng となっており、水槽水をそのまま使用した系の結果と比べると、各プロトコル間の総DNA量の差が少なくなっていました。
DNeasyのカラムのDNA保持能の検討
1-1の実験でInputされるDNA量が多い場合、アウトレット側から注入するプロトコルで抽出効率が低いことが分かりました。
この原因としてDNeasyのカラムのサンプルDNAで飽和して結合ステップの段階で保持されずに落ちてしまっている可能性があることが考えらます。
上記疑問を解消するため、2Lの水槽水と10倍希釈した1Lの水槽水をそれぞれ、高濃度群と低濃度群としてそれぞれでカラムの保持能力の検証をしました。
抽出はProtocol3(アウトレットから抽出液を逆噴射)の方法で行い、DNeasyのカラムを連結させて濾過することで、Upperで保持しきれずに通過したDNAをLowerでキャッチするようにしました。
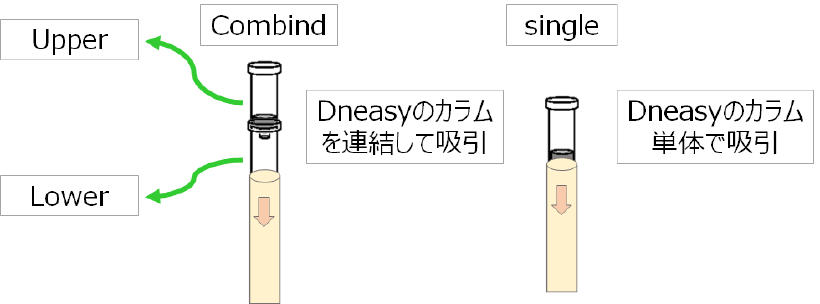
文中の説明より作成(多分こんな感じ?)
カラム保持能の検証結果
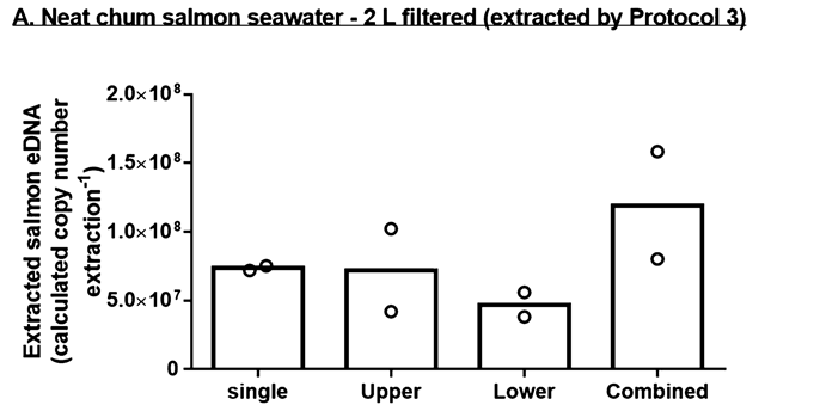
Fig.2を改変
水槽水を2Lろ過した系の場合、DNeasyのカラムを連結させてそれぞれで抽出したUpperとLowerは単体で抽出したsingleと同等の値を示しました。
つまり、Input量が多いとDNeasyのカラムのDNA保持能を超えてDNAが通過してしまう可能性があることを指してます。

Fig.2を改変
水槽水を10倍希釈して1Lろ過した系においても、Lowerで検出されていることから保持能を超えてしまっている可能性が考えられます。
長いので一度まとめます。
考察
フィルターに添加する溶解バッファーの量を増加(0.44mL → 2mL)させると、環境DNAの収量が劇的に増加しました。
環境DNAマニュアル記載の方法で収量が少なかった理由としては、図6A, Bのように溶解バッファーがフィルターと十分に接触せずうまく溶解ができていなかった可能性があることが挙げられます。

Fig.6 通常プロトコルの懸念要素に関する図
また、Protocol3のアウトレット側からの逆洗で抽出する方法で、環境DNAの収量がさらに増加(Fig.1 B)したことから、Sterivexカートリッジの細孔に閉じ込められて、うまく溶解されていない環境DNAが存在するかもしれません。
ですが、高濃度群では逆洗しても環境DNA量は増加しませんでした(図1A)。
これについては、カラム保持能の検証結果により説明ができるかもしれません。
Inputされる環境DNAの濃度が高い場合、連結したカラムの下側(Lower)で捕捉され(図2A)、連結した各カラムの環境DNA含有量は単一で抽出したカラムの含有量と類似していたことから、高濃度だとDNeasyのカラムの最大許容量を超えてしまっていたのではないかと考えられます。
抽出溶液のDNA量を計測したところ、DNeasyのカラムは数十μgのDNA保持能力があるにも関わらず、0.4ng * 1.2ngとDNeasyのカラムの規定容量よりも低い測定値が得られました。
この理由としては、環境サンプル中の未知の化合物や汚染物質がシリカ表面への非特異的に結合して、環境DNAの結合の競合または、結合を阻害を発生させている可能性があます。野外サンプル中には、プランクトンや藻類、糞便や分解された腐敗物などが豊富なため、これらのDNAや非特異的な結合によってDNeasyのカラムは保持能の限界を超えてしまう可能性があります。
これらの結果は、定量PCRによってサンプル中の環境DNA量を推定、またはメタバーコーディングで種構成を推定するためには、抽出段階で内部標準による抽出効率をモニタリングする必要性があることも示唆しています。
Part1は以上です。
免責事項
画像はジャーナルのCC BYに従う形で、記事で紹介したオープンアクセスの論文中の画像または、自身で作成したものを使用しています。
十分注意は払っていますが、本記事の情報・内容について保証されるものではありません。また、本記事の利用や閲覧によって生じたいかなる損害について責任を負いません。そして、本記事の情報は予告なく変更される場合がありますので、ご理解くださいますようお願い申し上げます。